1. 树空间的多维缩放(multidimensional scaling,MDS)可视化物种树和基因树的RF距离
- 简介
- 当系统发育拓扑冲突时,需要评估基因树与物种树的不一致程度,可使用树空间的多维缩放(multidimensional scaling,MDS)来对树的RF距离进行降维,并在指定维度可视化基因树与物种树之间的RF距离。
- 分析步骤
- 先使用R包ape 5.0计算树之间的成对Robinson-Foulds(RF)距离,然后对RF距离进行多维缩放(multidimensional scaling,MDS),最后绘制指定维度下的RF距离示意图。
- 结果
- MDS的二维图如下所示,图中的物种树,包括不同数据和不同软件的物种树(包括ASTRAL_tree,SCGs_ML,SNPs_ML,Plastome_ML树)用其他颜色标示,每个蓝色的点代表一棵基因树,颜色深浅代表支持率平均值的大小。
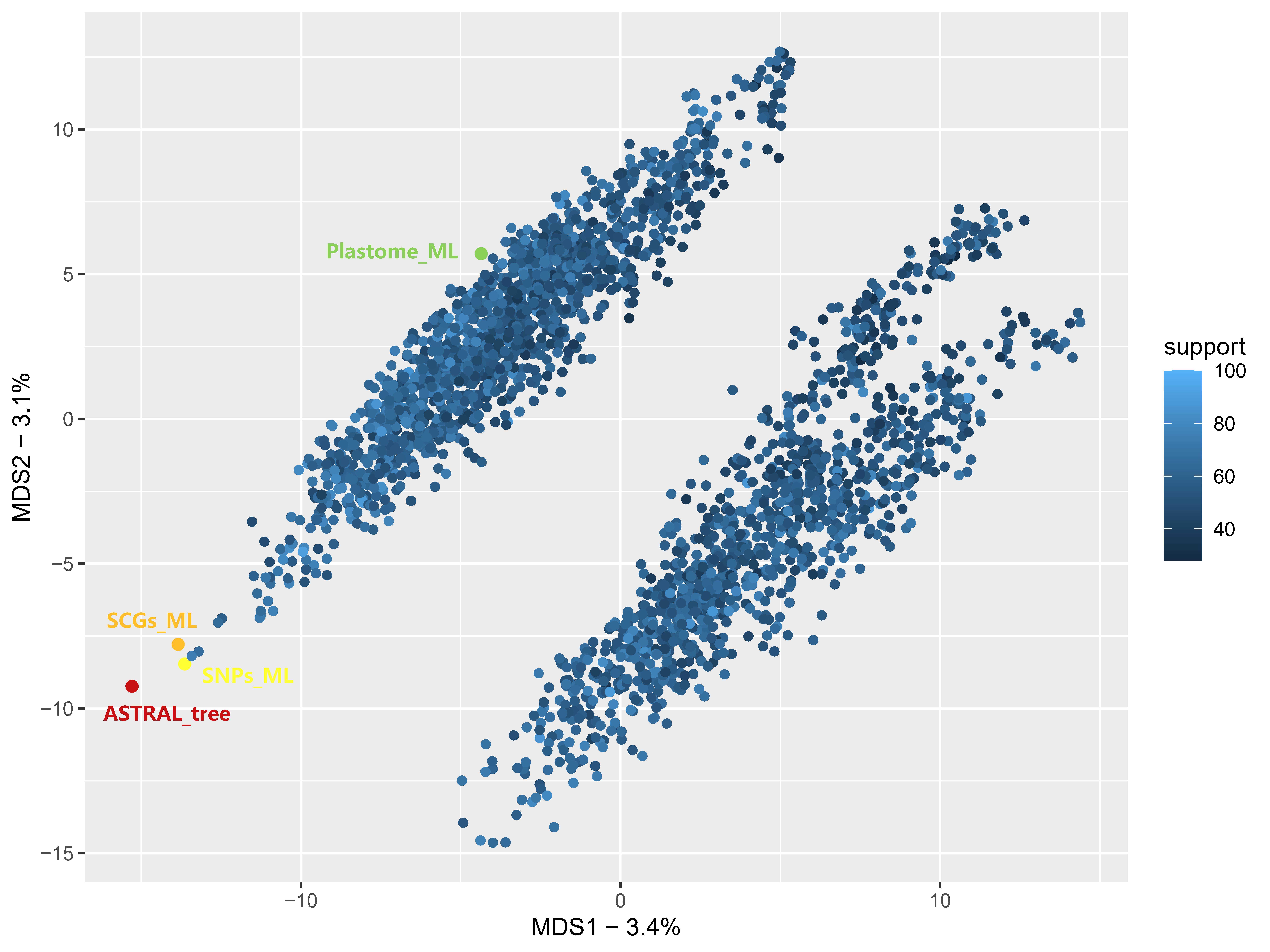
图 1. MDS二维图示例
2. 分析过程
- 输入文件
- species_genes.tre:物种树和基因树合并的树文件,可以合并多棵物种树(比如用不同算法和数据构建的树)。
- 用R包ape计算基因树成对间的Robinson-Foulds(RF)距离
1
2
3
4
5
| library(ape)
trees<-read.tree("species_genes.tre")
trees_dist<-dist.topo(trees, method = "PH85")
trees_matric<-as.matrix(trees_dist)
write.table (trees_matric, file ="RF_pairs.tsv", sep ="\t")
|
- RF_pairs.tsv文件是行和列都是tree1,tree2,…,treen等依次命名的树之间的RF距离矩阵。
- MDS多维缩放
1
2
3
4
5
| mds <- cmdscale(trees_dist, eig=TRUE, k=2)
mds.var.per <- round(mds$eig/sum(mds$eig)*100,1)
mds.values <- mds$points
mds.data <- data.frame(Sample=rownames(mds.values), X=mds.values[,1], Y=mds.values[,2])
write.table(mds.data,file="mds.tsv",sep="\t")
|
- mds.tsv文件示例。每棵树都有一行数据,这里是二维的,包含MDS1和MDS2两个值。用tree1,tree2,…,treen等依次命名,与species_genes.tre树文件中树的顺序一致。如果合并了物种树和基因树,可以依据顺序在mds.tsv中找到物种树对应的行。
1
2
3
| "Sample" "MDS1" "MDS2"
"tree1" "tree1" -15.2853071675022 -9.23665453167777
"tree2" "tree2" -13.8416686613703 -7.78853307096205
|
1
2
| library(MASS)
mds <- isoMDS(trees_dist, k=2)
|
- 绘图
1
2
3
4
5
6
7
| library(ggplot2)
ggplot(data=mds.data, aes(x=X, y=Y, label=Sample))+
geom_text() +
xlab(paste("MDS1 - ", mds.var.per[1], "%", sep=""))+
ylab(paste("MDS2 - ", mds.var.per[2], "%", sep=""))+
theme_bw()+
ggtitle("MDS plot using Euclidean distance")
|
或者
1
2
3
4
5
| mds <- cmdscale(trees_dist, eig=TRUE, k=2)
x <- mds$points[,1]
y <- mds$points[,2]
plot(x, y, xlab="Coordinate 1", ylab="Coordinate 2", main="Metric MDS", type="n")
text(mds$points, labels = rownames(trees_matric), cex = 0.6)
|
结果图保存成pdf后可以在pdf编辑器里把物种树对应的点用更大尺寸和其他颜色标示出来。
- 建议
- 还可以把每个基因树和物种树的支持率的平均值计算出来之后,用支持率给每棵树的MSD点上色,从而查看是否是离物种树越近的基因树的平均支持率越高。
- 也可以用基因树的平均支持率和基因树离物种树的RF距离画散点图,并做线性回归分析判断相关性。
3. references
- MDS paper:https://academic.oup.com/sysbio/article/67/3/400/4175806
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。




