clusterProfiler相关的博客共有三篇,共同食用,效果更好 :wink: :
- 博客富集分析:(三)clusterProfiler概述
- 博客富集分析:(四) clusterProfiler:不同物种的GO+KEGG富集分析
- 博客富集分析:(五)clusterProfiler:Visualization
1. 基因富集分析(gene set enrichment analysis, GSEA)
富集分析概述参考博客:富集分析:(一)概述。
2. clusterProfiler介绍
clusterProfiler是一个R包,是一个解释组学数据的通用富集工具许多基因集的功能注释和富集分析,以及富集分析结果的可视化。
2021年07月发布了clusterProfiler 4.0版本。
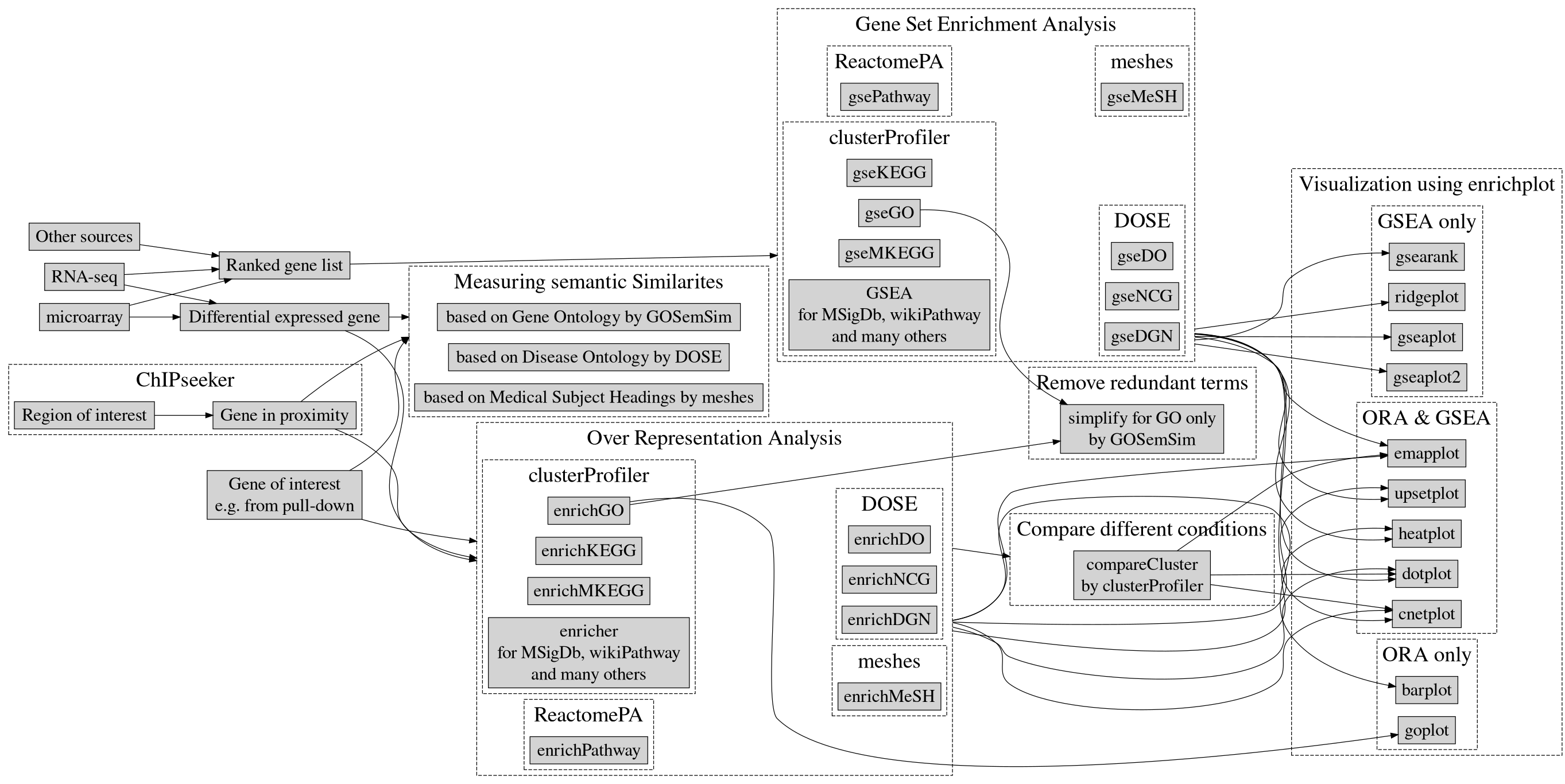
Figure 1. clusterProfiler function and workflow
图片来源: yulab’s book
3. clusterProfiler支持的基因集(gene sets)或基因通路数据库
- Gene Ontology(GO)
- Kyoto Encyclopedia of Genes and Genomes(KEGG)
- Disease Ontology(DO)
- Disease Gene Network(DisGeNET)
- Molecular Signatures Database(MSigDb)
- wikiPathways
… …
4. clusterProfiler功能 —— 富集分析(enrichment analysis)
4.1. 过表达分析(Over Representation Analysis, ORA)
过表达分析是用于采用超几何分布检验判断已知的生物功能或过程(例如GO/KEGG)在实验产生的基因列表(例如差异表达基因列表: differentially expressed genes, DEGs)中是否过表达(over-represented=enriched)的常用方法。
所有基因(或全基因组)作为背景总数N,被注释到已知基因集(例如GO/KEGG)某一子集的基因总数是M,差异表达的基因数量是n,差异表达基因中属于M的数量是k。
过表达分析就是用统计学的方法判断GO/KEGG的每一个子类中是否k/n显著高于M/N,显著高于就是这个子类过表达。
差异表达基因通常是前期通过比较转录组分析等手段获取的不同转录组间有表达差异的基因列表。
4.2. 基因集富集分析(Gene Set Enrichment Analysis, GSEA)
对于差异很大的基因,可以使用ORA分析,但差异较小的基因也可能具有意义,很多相关表型差异是通过一组基因中微小但一致的变化(small but consistent changes)来表现的。
基因集富集分析汇总一个基因集中每个基因的统计数据,可以检测预先定义的基因集(例如GO/KEGG)中所有基因以一种较小但协调的(small but coordinated)方式发生变化的情况。
基因根据表型进行排序,给定先验定义的基因集(例如共享相同GO/KEGG类别的基因),GSEA的目标是确定S的成员是随机分布在排序的基因列表L中,还是主要分布在顶部或者底部。
4.3. leading edge分析和核心富集基因(Leading edge analysis and core enriched genes)
包(clusterProfiler,DOSE,meshes,ReactomePA)支持Leading edge分析并可以提供GSEA分析中的核心富集基因core enriched genes结果。
- Leading edge分析可以获取:
- Tags显示对富集分数有贡献的基因的百分比
- List显示获得富集分数在列表中的位置
- Signal显示富集信号强度
5. clusterProfiler安装
启动R后输入下面命令安装
1 | if (!requireNamespace("BiocManager", quiet = TRUE)) |
6. clusterProfiler的GO和KEGG富集分析
clusterProfiler支持对许多ontology/pathway的hypergeometric test和gene set enrichment analyses,包括数据库GO,KEGG,DO,DisGeNET,MSigDb,wikiPathways。
使用支持的数据库时,需要检查分析样本是否在已有物种列表中。不在物种列表的物种数据,以及其他不在数据库列表的数据库,做分析时使用通用的富集分析(Universal enrichment analysis)模块。
分析前,先用命令library(clusterProfiler)载入clusterProfiler包。
6.1. 背景数据库选择
参考博客基因富集分析(gene set enrichment analysis, GSEA) —— clusterProfiler:不同物种的GO+KEGG富集分析。
6.2. GO富集分析
6.2.1. GO的ORA分析
6.2.1.1. 输入文件
ORA分析的输入文件是gene ID list,比如差异表达分析(DESeq2)获得的差异表达基因列表,保存为内容为一列数据的文本文件gene.list,数据内容可以是OrgDb支持的任意ID类型(常用的都支持,ENSEMBL,ENTREZID,GENETYPE,GO,PFAM),具体参考ID。
分析的是列表中基因在GO/KEGG各个子分类单元是否被过度代表还是代表不足。
6.2.1.2. bitr格式转换
如果需要,也可以用bitr功能函数实现各种ID格式的转换。
1 | data <- read.table("gene.list",header=FALSE) #单列基因名文件 |
6.2.1.3. GO分类
groupGO()函数可以基于GO在指定水平范围内的分布进行基因分类。
1 | data <- read.table("gene.list",header=F) #读取gene ID list,内容为一列ENSEMBL格式的基因ID名称 |
6.2.1.4. ORA分析
- 读取输入文件
- 读取基因ID的list文件
- 这些ID会在enrichGO的分析中被自动去重,所以文件是否有重复结果都一样。
1 | data <- read.table("gene.list",header=F) #读取gene ID list,内容为一列ENSEMBL格式的基因ID名称 |
- 支持物种的标准分析 enrichGO()
1 | ego <- enrichGO(gene = genes, # list of entrez gene id |
提供注释文件go_annotation.txt的非模式物种分析 enricher()
1
2
3
4go_anno <- read.table("go_annotation.txt",header = T,sep = "\t")
go2gene <- go_anno[, c(2, 1)]
go2name <- go_anno[, c(2, 3)]
ego <- enricher(genes, TERM2GENE = go2gene, TERM2NAME = go2name, pAdjustMethod = "BH",pvalueCutoff = 0.05, qvalueCutoff = 0.2)ego结果文件解释
- ONTOLOGY:CC BP MF
- ID: Gene Ontology数据库中唯一的标号信息
- Description :Gene Ontology功能的描述信息
- GeneRatio:差异基因中与该Term相关的基因数与整个差异基因总数的比值
- BgRatio:所有( bg)基因中与该Term相关的基因数与所有( bg)基因的比值
- pvalue: 富集分析统计学显著水平,一般情况下, P-value < 0.05 该功能为富集项
- p.adjust 矫正后的P-Value
- qvalue:对p值进行统计学检验的q值
- geneID:与该Term相关的基因
- Count:与该Term相关的基因数
- 输出结果保存为csv表格
1
write.table(as.data.frame(ego),"go_enrich.csv",sep="\t",row.names =F,quote=F) #保存到文件go_enrich.csv。其中as.data.frame(ego)把ego对象转换成数据框dataframe
6.2.1.5. 结果整理和筛选
- 如果没使用keyType参数,可以在得到结果后使用
- 如果没使用readable=True参数,可以在得到结果后用函数setReadable()将GeneID转换为symbol。
ego <- setReadable(ego, OrgDb = org.At.tair.db) - 函数dropGO可以移除enrichGO结果中特定的GO term或GO level
- 函数gofilter()可以将结果限定在特定的GO level
- 函数simplify可以去除冗余,
ego_rm <- simplify(ego, cutoff=0.7, by="p.adjust", select_fun=min)
6.2.2. GO的GSEA分析
GSEA分析通过置换检验来计算p值
6.2.2.1. GSEA的输入文件
- GSEA分析的输入文件是一个基因排序列表,有三个要点:
- numeric vector:倍数变化或者其他类型的数字变量,比如差异表达分析里的logFC值
- named vector:每个数字对应的gene ID命名
- sorted vector;数字应该以降序排序
即包含两列,一列基因ID名称,一列数据,并以数据降序排序。
- 获取输入文件的示例:
1 | d <- read.csv(your_csv_file) |
1 | > head(mydata,3) |
6.2.2.2. GSEA分析
读取输入文件
1
2
3
4data <- read.csv("geneList.csv")
geneList <- d[,2]
names(geneList) <- as.character(d[,1])
geneList <- sort(geneList, decreasing = TRUE)支持物种的标准分析 gseGO()
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26gsego <- gseGO(geneList = geneList,
OrgDb = org.At.tair.db,
ont = "BP", # "BP", "MF", "CC", "ALL"。GO三个子类。如果选ALL,会同时获得三个子类的结果(相当于单独做三个子类的结果合并在一起),结果中增加一列ONTOLOGY为子类。
keyType = "SYMBOL", # 输入基因的类型,命令keytypes(org.Hs.eg.db)会列出可用的所有类型;
nPerm = 1000, # permutation numbers 置换次数
minGSSize = 100, # 富集基因数量的下限
maxGSSize = 500, # 富集基因数量的上限
pvalueCutoff = 0.05,
verbose = FALSE, # 不输出结果
by = "fgsea") # 选项fgsea或DOSE
head(gsego,1);dim(gsego)
ID Description setSize enrichmentScore
GO:0003674 GO:0003674 molecular_function 323 0.9462411
NES pvalue p.adjust qvalues rank
GO:0003674 1.716837 0.000999001 0.000999001 NA 579
leading_edge
GO:0003674 tags=100%, list=11%, signal=95%
core_enrichment
GO:0003674 CG15892/CG15136/CG4983/CG43851/…
[1] 53 11
head(data.frame(gsego$ID,gsego$Description))
gsego.ID gsego.Description
1 GO:0003674 molecular_function
2 GO:0005488 binding提供注释文件go_annotation.txt的非模式物种分析 GSEA()
1
2
3
4data <- read.table("go_annotation.txt",header = T,sep = "\t",quote="")
go2gene <- data[, c(2, 1)]
go2name <- data[, c(2, 3)]
gsego <- GSEA(genes, TERM2GENE = go2gene, TERM2NAME = go2name)gsego结果文件解释
- ID: Gene Ontology数据库中唯一的标号信息
- Description:Gene Ontology功能的描述信息
- setSize
- enrichmentScore:富集分数
- NES
- pvalue: 富集分析统计学显著水平,一般情况下, P-value < 0.05 该功能为富集项
- p.adjust:矫正后的P-Value
- qvalues:对p值进行统计学检验的q值
- rank
- leading_edge
- core_enrichment
6.2.3. GO富集结果可视化
goplot(ego)简单可视化结果为有向无环图。
6.3. KEGG富集分析
clusterProfiler通过KEGG数据库的API来获取KEGG的注释信息,包括一个物种所有基因对应的pathway注释文件,比如人的:http://rest.kegg.jp/link/hsa/pathway;和pathway对应的描述信息,比如人的:http://rest.kegg.jp/list/pathway/hsa。
6.3.1. 支持的物种
- clusterProfiler包支持的物种
只需要将物种缩写输入给clusterProfiler,clusterProfiler包支持自动联网调取kegg注释物种列出物种的pathway注释信息,网站可以查看物种列表和缩写,或者用clusterProfiler包提供search_kegg_organism()函数来帮助搜索支持的生物。
1 | search_kegg_organism('osa', by='kegg_code') #查询kegg_code为osa的记录,水稻 |
- 用户提供KEGG pathway注释数据
如果分析的物种不支持自动调取,可以自己做KEGG pathway注释后提供注释文件,比如interproscan、eggnog-mapper等软件的注释结果,整理成clusterProfiler支持的输入格式即可。
clusterProfiler需要导入的KEGG pathway注释文件pathway_annotation.txt的格式如下:
| GeneID | Pathway | Path_Description |
|---|---|---|
| 1 | ko:00001 | spindle |
| 2 | ko:00002 | mitotic spindle |
| 3 | ko:00003 | kinetochore |
data.frame格式,包含三列,第一列为Gene ID,第二列为 KEGG Pathway ID,第三列为Path_Description,顺序无要求。
6.3.2. KEGG pathway的ORA分析
6.3.2.1. KEGG输入ID的格式转换
ID转换函数
1 | library(clusterProfiler) |
6.3.2.2. KEGG pathway的ORA分析
输入文件与GO的ORA分析输入文件一样。
- 导入输入文件
1 | data <- read.table("gene.list",header=F) #读取gene ID list,内容为一列ENSEMBL格式的基因ID名称 |
- 支持物种的标准分析 enrichKEGG()
1 | kk <- enrichKEGG(gene = genes, |
输入的ID类型默认是kegg gene id,也可以是ncbi-geneid或ncbi-proteinid或者uniprot等,可以通过上一步格式转换转换ID类型。
organism对应物种的三字母缩写,其他参数与GO的ORA分析参数一致。
不像enrichGO()函数有readable()参数,enrichKEGG()函数没有这个参数,当待分析物种在OrgDb数据库中可用时,可以用setReadable()函数把gene ID转换成gene Name。
- 提供注释文件pathway_annotation.txt的非模式物种分析 enricher()
1 | pathway_anno <- read.table("pathway_annotation.txt",header = T,sep = "\t") |
- 结果输出到csv文件
write.table(as.data.frame(kk),"KEGG_enrich.csv",sep="\t",row.names =F,quote=F)#保存到文件KEGG_enrich.csv。其中as.data.frame(kk)把kk对象转换成数据框dataframe
6.3.3. KEGG pathway的GSEA分析
读取输入文件
1
2
3
4data <- read.csv("geneList.csv")
geneList <- d[,2]
names(geneList) <- as.character(d[,1])
geneList <- sort(geneList, decreasing = TRUE)支持物种的标准分析 gseKEGG()
1 | kks <- gseKEGG(geneList = geneList, |
- 提供注释文件pathway_annotation.txt的非模式物种分析 GSEA()
1
2
3
4data <- read.table("pathway_annotation.txt",header = T,sep = "\t")
go2gene <- data[, c(2, 1)]
go2name <- data[, c(2, 3)]
kks <- GSEA(gene, TERM2GENE = go2gene, TERM2NAME = go2name)
6.3.4. KEGG module的ORA分析
KEGG Module是手动定义的功能单元的集合。在某些情况下,KEGG 模块有更直接的解释。
1 | mkk <- enrichMKEGG(gene = gene, |
6.3.5. KEGG module的GSEA分析
1 | mkk2 <- gseMKEGG(geneList = geneList, |
6.3.6. KEGG pathways富集结果的可视化
enrichplot包可以实现几种方法,可以用在GO,KEGG,MSigDb等基因集注释上。
browseKEGG()函数会打开网络浏览器突出显示富集的基因的KEGG通路,用hsa04110基因举例:
browseKEGG(kk, 'hsa04110')pathview::pathview()可视化富集的KEGG通路
1
2
3
4
5library("pathview")
hsa04110 <- pathview(gene.data = geneList,
pathway.id = "hsa04110",
species = "hsa",
limit = list(gene=max(abs(geneList)), cpd=1))
6.4. 通用的富集分析(Universal enrichment analysis)
除GO,KEGG基因集外,clusterProfiler还支持WikiPathways,Reactome,Disease,MeSH等数据库的富集分析。
虽然clusterProfiler支持对许多ontology/pathway的hypergeometric test和gene set enrichment analyses,但是还有很多数据,包括不支持的物种、不支持的ontologies/pathways或自定义注释等。
clusterProfiler提供了用于hypergeometric test的enricher()函数和用于基因集富集分析的GSEA()函数,用于接受用户定义的注释。
另外两个参数TERM2GENE和TERM2NAME:
- TERM2GENE是一个必需的data.frame,第一列为term ID,第二列为对应映射基因;
- TERM2NAME是一个可选的data.frame,第一列为term ID,第二列为对应term name。
在GO和KEGG富集分析的用户自行提供注释文件的分析部分,使用的enricher()和GSEA()函数。
7. references
- GSEA wiki: https://en.wikipedia.org/wiki/Gene_set_enrichment_analysis
- enrichment: https://www.jianshu.com/p/47b5ea646932
- clusterProfiler github: https://github.com/YuLab-SMU/clusterProfiler
- clusterProfiler paper: https://www.cell.com/the-innovation/fulltext/S2666-6758(21)00066-7?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS2666675821000667%3Fshowall%3Dtrue
- clusterProfiler book: http://yulab-smu.top/biomedical-knowledge-mining-book/index.html
- clusterProfiler manual: https://bioconductor.org/packages/devel/bioc/manuals/clusterProfiler/man/clusterProfiler.pdf
- clusterProfiler ducumentation: https://guangchuangyu.github.io/software/clusterProfiler/documentation/
- clusterProfiler blog: https://guangchuangyu.github.io/2016/01/go-analysis-using-clusterprofiler/
- tutorial: https://www.cnblogs.com/jessepeng/p/12159139.html
- 函数simplify: http://guangchuangyu.github.io/2015/10/use-simplify-to-remove-redundancy-of-enriched-go-terms/
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。


