1. 直系同源(orthology)推断
1.1. 直系同源推断
直系同源(orthology)是指直系同源基因的直系同源这种属性,相关的概念可以参考博文-同源性相关概念。
直系同源推断(orthology inference)常常在多个物种间进行,通常是获取多个物种的直系同源群(orthogroups)和直系同源群间的关系。
直系同源基因判定的方法有两种:
- 一种是将物种的直系同源基因两两比较,不同物种的每一对都是直系同源,但存在基因重复,使得用这种方法找到的同源关系是不具有传递性的(A和B是同源的,B和C是同源的,但是A和C不是同源的)。multiparanoid和OMA方法都是这个原理。所以需要每个同源基因都在多个集合中存在才行,进行两两比较。
- 另一种是识别完整的正交群,一个同源组orthogroup包含所有物种共同祖先基因,同时包含直系/旁系同源基因,OrthoMCL是这个原理,它是通过正交组做推断,用BLAST计算多个物种序列之间的序列相似性得分,然后用MCL聚类算法,识别该数据集中高度相似的序列组。
这篇博文主要介绍一个新工具PlantTribes2。
2. PlantTribes2介绍
PlantTribes 是用于比较植物基因组学的自动化基因家族分析流程的集合。PlantTribes通过模块对植物基因组中完整蛋白质序列聚类来进行比较进化研究。 模块功能包括:将从头组装的转录本处理为推断的编码序列及其相应的氨基酸翻译序列,局部组装目标基因家族,推断直系同源基因,并进行基因家族多序列比对和构建相应的系统发育树,以及估算一组基因序列的旁系/直系同源的成对同义/非同义替换率。
3. PlantTribes_scaffold数据库
PlantTribes2 (v1.0.4) 基于过去发表的系统基因组学研究提供了几个植物基因家族scaffolds数据库,包含了预先构建的多序列比对和orthogroups,下载网址是:http://bigdata.bx.psu.edu/PlantTribes_scaffolds/data/
- 单子叶植物数据库:12Gv1.0(代表12个物种的scaffolds)
- 被子植物数据库的四个版本:22Gv1.1, 26Gv1.0, 26Gv2.0, and 37Gv1.0
4. PlantTribes2的模块
PlantTribes2一共有7个模块,每个模块是独立的功能,也可以联合使用。
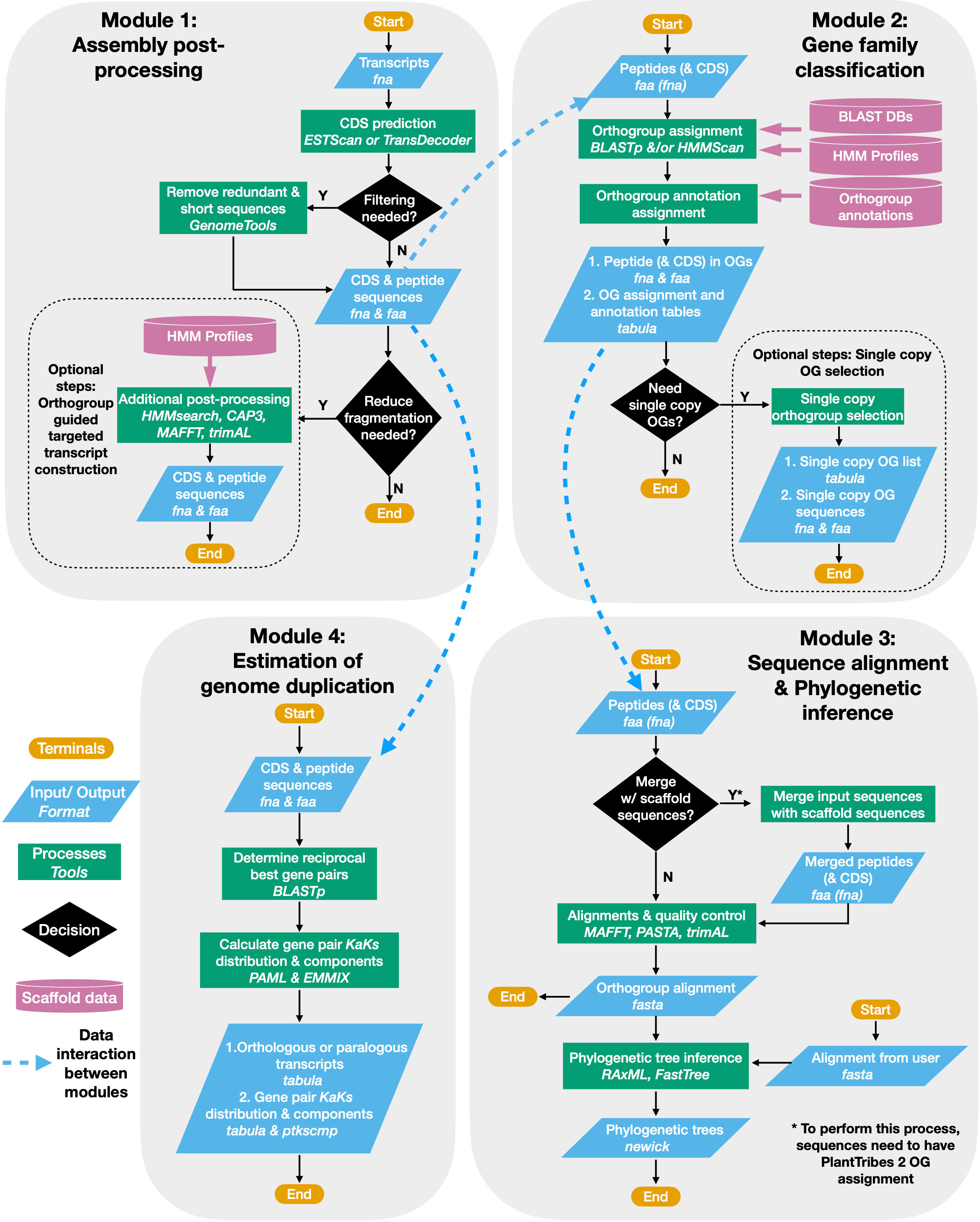
Figure 1. PlantTribes2 analysis workflow
from PlantTribes2 analysis workflow
4.1. AssemblyPostProcessor模块
- 运行 ESTScan 编码区域预测
PlantTribes/pipelines/AssemblyPostProcessor --transcripts transcripts.fasta --prediction_method estscan --score_matrices /path/to/score/matrices/Arabidopsis_thaliana.smat
4.2. GeneFamilyClassifier模块
- 用22Gv1.1 scaffolds,orthomcl 聚类法 和blastp 分类器运行
PlantTribes/pipelines/GeneFamilyClassifier --proteins proteins.fasta --scaffold 22Gv1.1 --method orthomcl --classifier blastp
4.3. PhylogenomicsAnalysis模块 (legacy pipeline)
- 用22Gv1.1 scaffolds,orthomcl 聚类法 和raxml建树法运行
PlantTribes/pipelines/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --add_alignments --tree_inference raxml
4.4. GeneFamilyIntegrator模块
- 用22Gv1.1 scaffolds,orthomcl 聚类法运行
GeneFamilyIntegrator --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl
4.5. GeneFamilyAligner模块
- 用22Gv1.1 scaffolds,orthomcl 聚类法和mafft比对软件运行
GeneFamilyAligner --orthogroup_faa integratedGeneFamilies_dir --alignment_method mafft
4.6. GeneFamilyPhylogenyBuilder模块
- 用22Gv1.1 scaffolds,orthomcl 聚类法和fastree建树软件运行
GeneFamilyPhylogenyBuilder --orthogroup_aln geneFamilyAlignments_dir/orthogroups_aln --tree_inference fasttree
4.7. KaKsAnalysis模块
- 进行旁系同源分析
KaKsAnalysis --coding_sequences_species_1 species1.fna --proteins_species_1 species1.faa --comparison paralogs --num_threads 4
5. PlantTribes2安装
- conda安装:PhylogenomicsAnalysis模块无法通过conda安装
- AssemblyPostProcessor模块:
conda install bioconda::plant_tribes_assembly_post_processor - GeneFamilyAligner模块:
conda install bioconda::plant_tribes_gene_family_aligner - GeneFamilyClassifier模块:
conda install bioconda::plant_tribes_gene_family_classifier - GeneFamilyIntegrator模块:
conda install bioconda::plant_tribes_gene_family_integrator - GeneFamilyPhylogenyBuilder模块:
conda install bioconda::plant_tribes_gene_family_phylogeny_builder - KaKsAnalysis模块:
conda install bioconda::plant_tribes_kaks_analysis
- 下载安装
- 确保依赖软件都已安装
- 下载PlantTribes的github库:
git clone https://github.com/dePamphilis/PlantTribes.git - PlantTribes的可执行文件在pipelines子目录下
6. PlantTribes2快速使用
6.1. 标准运行
1 | nohup AssemblyPostProcessor --transcripts Trinity.fasta --prediction_method transdecoder --scaffold /path/to/PlantTribes_scaffolds/22Gv1.1 --method orthofinder --dereplicate --min_length 100 --num_threads 12 & # 预测转录本的CDS区域 |
6.2. focus on目标基因的运行
1 | 预测转录本的CDS区域,并组装RRRgenes_orthogroups.ids文件中指定的orthogroups家族的基因。 |
个人经验:这种运行方式focus on目标基因,去掉了GeneFamilyClassifier模块的使用,计算量小很多,能很快的得到目标基因的结果。虽然能够尽可能地拼接出目标基因的转录本,但是可能包含更多假阳性结果。
推荐:在使用AssemblyPostProcessor模块并且–gene_family_search参数指定目标基因id的list,获得转录本CDS区域后,就按照标准运行流程依次使用GeneFamilyClassifier模块、GeneFamilyIntegrator模块、GeneFamilyAligner模块、GeneFamilyPhylogenyBuilder模块来进行直系同源转录本的推断,结果更可靠。
同时建议对GeneFamilyAligner模块获得的align比对好的序列进行手动和肉眼检查,看是否有假阳性的结果(明显比对不上的转录本序列被放进了结果)。
7. PlantTribes2运行的详细参数
用de novo转录组组装得到的Trinity.fasta文件来演示PlantTribes2运行流程。
7.1. 准备数据
- 下载你想用的PlantTribes_scaffold数据库,从数据库网址(http://bigdata.bx.psu.edu/PlantTribes_scaffolds/data/)中选择一个数据库下载,检查并解压缩`wget http://bigdata.bx.psu.edu/PlantTribes_scaffolds/data/22Gv1.1.tar.bz2`,`md5sum 22Gv1.1.tar.bz
,tar -xjvf 22Gv1.1.tar.bz2`。 - 下载Arabidopsis_thaliana.smat矩阵文件,
wget https://github.com/dePamphilis/PlantTribes/raw/master/test/Arabidopsis_thaliana.smat.gz && gzip -d Arabidopsis_thaliana.smat.gz
7.2. 组装后处理模块:AssemblyPostProcessor Pipeline
AssemblyPostProcessor Pipeline 对de novo组装的转录本进行蛋白编码区域预测,获得CDS区域和去除冗余后的转录组序列,用于后续分析。
7.2.1. 运行
- 用ESTScan预测coding region
PlantTribes/pipelines/AssemblyPostProcessor --transcripts Trinity.fasta --prediction_method estscan --score_matrices /path/to/score/matrices/Arabidopsis_thaliana.smat --strand_specific --dereplicate --min_length 200 --num_threads 12
- 用TransDecoder预测coding region
PlantTribes/pipelines/AssemblyPostProcessor --transcripts Trinity.fasta --prediction_method transdecoder --strand_specific --dereplicate --min_length 200 --num_threads 12- 输出文件包括:
- assemblyPostProcessing_dir/transcripts.cds
- assemblyPostProcessing_dir/transcripts.pep
- assemblyPostProcessing_dir/transcripts.cleaned.cds
- assemblyPostProcessing_dir/transcripts.cleaned.pep
- assemblyPostProcessing_dir/transcripts.cleaned.nr.cds:用BLASTp(evalue=1e-5)把transcripts跟NCBI的NR库比对去除非植物的污染序列之后的序列。
- assemblyPostProcessing_dir/transcripts.cleaned.nr.pep:用BLASTp(evalue=1e-5)把transcripts跟NCBI的NR库比对去除非植物的污染序列之后的序列。
- 用TransDecoder预测coding region,并指定目标基因家族选项
PlantTribes/pipelines/AssemblyPostProcessor --transcripts Trinity.fasta --prediction_method transdecoder --strand_specific --dereplicate --min_length 200 --num_threads 12 --gene_family_search targetOrthos.ids --scaffold 22Gv1.1 --method orthofinder- 输出文件除了上面的,还包括:
- assemblyPostProcessing_dir/targeted_gene_family_assemblies.stats
- assemblyPostProcessing_dir/targeted_gene_family_assemblies/目录下包括以targetOrthos.ids命名的fasta, fna, 和 faa文件。(faa是氨基酸序列,其他两个是核苷酸序列)
- notes:实际运行发现,使用了
--gene_family_search指定目标基因家族后,会额外组装目标基因家族的转录本,获得更长的目标基因家族的转录本,以及更多转录本数量的转录组数据。7.2.2. 参数
- 必需参数:
--transcripts:de novo 转录组组装的 fasta 文件--prediction_method:coding regions的预测方法,ESTScan方法(参数:estscan)或者TransDecoder方法(参数:transdecoder)。--score_matrices:如果预测方法选的ESTScan则为必需参数,(i.e. Arabidopsis_thaliana.smat, Oryza_sativa.smat, Zea_mays.smat)。
- 目标基因家族组装的参数:
--gene_family_search:目标orthogroup identifiers的list文件。需要”–scaffold” and “–method”两个参数。如果有目标基因想要分析(比如有个课题分析RRR基因),那么一定要指定以得到更有针对性的转录本。scaffold数据库文件里的22Gv1.1/annot/orthofinder.list保存所有orthogroup IDs(如果使用orthofinder的话)。目标list文件可以通过目标基因的拟南芥或者其他21个物种的ID来搜索orthofinder.list文件,搜索到的第一列(数字)即为目标orthogroup IDs。--scaffold:Orthogroups或者gene families proteins scaffold文件。用绝对路径指定。eg. /home/scaffolds/22Gv1.1--method:蛋白聚类方法。GFam:gfam;OrthoFinder:orthofinder;OrthoMCL:orthomcl;其他非PlantTribes方法:methodname。--gap_trimming:在alignments里移除gappy sites,默认0.1,代表移除含有90% gaps的sites。--min_coverage:在orthogroup trimmed蛋白多序列比对中最小序列覆盖度。默认0.0。推荐设置至少覆盖平均backbone orthogroup gene models。目标基因家族组装汇总统计输出文件中有细节说明。
- 其他参数:
--strand_specific:如果转录组是链特异性的de novo组装的,则加上这个参数,用strand_specific库运行。--dereplicate:在预测的蛋白编码区域移除重复序列。--min_length:预测的蛋白编码区域的最短序列长度。--num_threads:线程,CPU数量,默认1。
7.3. 基因家族分类模块:GeneFamilyClassifier Pipeline
GeneFamilyClassifier Pipeline 对预测得到的蛋白编码序列进行基因家族分类,输入文件是AssemblyPostProcessor Pipeline得到的转录本蛋白序列,使用PlantTribes搜集的22Gv1.1作为scaffolds数据库。
7.3.1. 运行
- 用22Gv1.1 scaffolds数据库,orthomcl 聚类法 和hmmscan分类器运行
PlantTribes/pipelines/GeneFamilyClassifier --proteins assemblyPostProcessing_dir/transcripts.cleaned.nr.pep --scaffold /path/to/22Gv1.1 --method orthomcl --classifier hmmscan --num_threads 10- 输出文件包括:
- geneFamilyClassification_dir/proteins.hmmscan.22Gv1.1
- geneFamilyClassification_dir/proteins.hmmscan.22Gv1.1.bestOrthos
- geneFamilyClassification_dir/proteins.hmmscan.22Gv1.1.bestOrthos.summary
- 参数
--classifier both同时使用hmmscan和blastp
- 输出文件包括
- geneFamilyClassification_dir/proteins.blastp.22Gv1.1
- geneFamilyClassification_dir/proteins.blastp.22Gv1.1.bestOrthos
- geneFamilyClassification_dir/proteins.hmmscan.22Gv1.1
- geneFamilyClassification_dir/proteins.hmmscan.22Gv1.1.bestOrthos
- geneFamilyClassification_dir/proteins.both.22Gv1.1.bestOrthos
- geneFamilyClassification_dir/proteins.both.22Gv1.1.bestOrthos.summary
- 按分类单元自主选择单拷贝/低拷贝基因家族
- 方法一:加上参数
--single_copy_custom - 方法二:加上参数
--single_copy_taxa 20 --taxa_present 21 - 输出文件除了上面的,还包括:
- geneFamilyClassification_dir/proteins.both.22Gv1.1.bestOrthos.summary.singleCopy
- 生成基因家族fasta文件
- 加上参数
--orthogroup_fasta --coding_sequences assemblyPostProcessing_dir/transcripts.cleaned.nr.cds - 输出文件除了上面,还包括:
- geneFamilyClassification_dir/orthogroups_fasta - transcriptome assembly orthogroup fasta directory
- geneFamilyClassification_dir/single_copy_fasta - transcriptome assembly single/low copy orthogroup fasta directory
7.3.2. 参数
- 必需参数:
--proteins:AssemblyPostProcessor Pipeline得到的氨基酸序列。--scaffold:Orthogroups或者gene families proteins scaffold文件。用绝对路径指定。eg. /home/scaffolds/22Gv1.1--method:蛋白聚类方法。GFam:gfam;OrthoFinder:orthofinder;OrthoMCL:orthomcl;其他非PlantTribes方法:methodname。--classifier:蛋白分类方法。BLASTP:blastp;HMMScan:hmmscan;BLASTP+HMMScan:both。BLASTP更快,HMMScan对远源homologs更敏感,both则更全面。
- 其他参数:
--config_dir:【optional】设置scaffold的配置文件的目录的绝对路径。例如,/home/config/22gv1.1。如果不设置这个参数,默认配置文件所在目录为~home/config。--num_threads:线程,CPU数量,默认1。用在设置BLASTP,HMMScan,MAFFT。--super_orthogroups:SuperOthogroups MCL聚类,所有orthogroups对之间的blastp e-value矩阵。If minimum e-value:min_evalue (default);If average e-value: avg_evalue。--single_copy_custom:自定义选择单拷贝orthogroup的配置文件。与–single_copy_taxa不兼容。需要绝对路径,默认。--single_copy_taxa:在orthogroup中需要的最少的单拷贝taxa数量,与–single_copy_custom不兼容。--taxa_present:在单拷贝orthogroup中需要的最少taxa数量,与–single_copy_taxa共用。--orthogroup_fasta:生成orthogroup fasta文件,需要–coding_sequences生成CDS orthogoup fasta序列。--coding_sequences:对应的coding sequences (CDS) fasta 序列。
7.4. PhylogenomicsAnalysis模块 (过时的流程,legacy pipeline)
- 整合scaffolds基因家族序列和分类好的转录本序列
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --orthogroup_fna- 输出文件:
- phylogenomicsAnalysis_dir/orthogroups_fasta/ - orthogroup fasta directory
- 用MAFFT L-INS-i模式生成多序列比对MSA文件
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --create_alignments
- 把未比对的转录组组装序列加入预先计算的scaffold基因家族多序列比对MSA文件
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --add_alignments
- 用PASTA方法(适合更大的数据集)生成多序列比对MSA文件
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --pasta_alignments- 输出文件:
- phylogenomicsAnalysis_dir/orthogroups_fasta/ - orthogroup fasta directory
- phylogenomicsAnalysis_dir/orthogroups_aln/ - orthogroup multiple sequence alignments directory
- 用RAxML构建ML基因家族系统树
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --pasta_alignments --tree_inference raxml
- 用FastTree构建approximately-maximum-likelihood基因家族树(更快)
PlantTribes/legacy/PhylogenomicsAnalysis --orthogroup_faa geneFamilyClassification_dir/orthogroups_fasta --scaffold 22Gv1.1 --method orthomcl --pasta_alignments --tree_inference fasttree- 输出文件:
- phylogenomicsAnalysis_dir/orthogroups_fasta/ - orthogroup fasta directory
- phylogenomicsAnalysis_dir/orthogroups_aln/ - orthogroup multiple sequence alignments directory
- phylogenomicsAnalysis_dir/orthogroups_tree/ - orthogroup phylogenetic trees directory
7.5. GeneFamilyIntegrator模块
- 用22Gv1.1 scaffolds,orthomcl 聚类法,整合分类好的转录组序列和scaffold基因家族序列
PlantTribes/pipelines/GeneFamilyIntegrator --orthogroup_fasta geneFamilyClassification_dir/orthogroups_fasta/ --scaffold 22Gv1.1 --method orthomcl- 输出文件:integratedGeneFamilies_dir/ - orthogroup fasta directory
- 参数
--orthogroup_fasta:指定包含基因家族分类orthogroups fasta files的目录。--scaffold:指定scaffold文件。--method:蛋白聚类方法。GFam:gfam;OrthoFinder:orthofinder;OrthoMCL:orthomcl;其他非PlantTribes方法:methodname。
7.6. GeneFamilyAligner模块
7.6.1. 运行
- 用MAFFT L-INS-i模式生成基因家族多序列比对MSA文件
PlantTribes/pipelines/GeneFamilyAligner --orthogroup_faa integratedGeneFamilies_dir --alignment_method mafft
- 用PASTA软件(适合更大的数据集)生成基因家族多序列比对MSA文件
PlantTribes/pipelines/GeneFamilyAligner --orthogroup_faa integratedGeneFamilies_dir --alignment_method pasta --pasta_script_path /path/to/pasta-code/pasta/run_pasta.py- 输出文件:
- geneFamilyAlignments_dir/orthogroups_aln_faa - orthogroup multiple sequence alignments directory
7.6.2. 参数
- 必需参数
--orthogroup_faa:指定包含基因家族分类orthogroups fasta files的目录。--alignment_method:多序列比对MSA方法。MAFFT:mafft;PASTA:pasta。
- codon alignments参数
--codon_alignments:构建orthogroup multiple codon比对,对应基因家族分类。orthogroups CDS文件需在orthogroups蛋白fasta文件同一目录下。
- MSA质控参数
--automated_trimming:用trimAl进行trims。--gap_trimming:在alignments中移除gappy位点。--remove_sequences:在alignments中移除gappy序列。--iterative_realignment:--remove_sequences移除时迭代的最大次数。
- 其他参数
--num_threads:用于MAFFT或PASTA的线程数,默认是1。--max_memory:最大可用内存。--pasta_iter_limit:PASTA算法迭代的最大次数。--pasta_script_path:用于PASTA的脚本run_pasta.py的位置。
7.7. GeneFamilyPhylogenyBuilder模块
7.7.1. 运行
- 用RAxML构建ML基因家族系统树
PlantTribes/pipelines/GeneFamilyPhylogenyBuilder --orthogroup_aln geneFamilyAlignments_dir/orthogroups_aln_faa --scaffold 22Gv1.1 --method orthomcl --tree_inference raxml- 输出文件:
- geneFamilyPhylogenies_dir/phylip_aln/ - orthogroup phylip multiple sequence alignments directory
- geneFamilyPhylogenies_dir/orthogroups_tree/ - orthogroup phylogenetic trees directory
- 用FastTree构建approximately-maximum-likelihood基因家族树(更快)
PlantTribes/pipelines/GeneFamilyPhylogenyBuilder --orthogroup_aln geneFamilyAlignments_dir/orthogroups_aln_faa --tree_inference fasttree- 输出文件:
- geneFamilyPhylogenies_dir/orthogroups_tree/ - orthogroup phylogenetic trees directory
7.7.2. 参数
GeneFamilyPhylogenyBuilder模块建树默认是设定外类群的,通常设定Ambtr1.0.27或Phypa1.6等为外类群,所以获得的树结果可直接用。
- 必需参数
--orthogroup_aln:指定包含基因家族orthogroups alignment files的目录。--tree_inference:系统发育树推断方法。RAxML:raxml;FastTree:fasttree。
- 其他参数
--scaffold:orthogroups或者基因家族蛋白scaffold。可以用绝对路径指定scaffold文件。--method:蛋白聚类方法。GFam:gfam;OrthoFinder:orthofinder;OrthoMCL:orthomcl;其他非PlantTribes方法:methodname。--config_dir:包含默认配置文件的绝对路径。--max_orthogroup_size:orthogroup比对的最大序列数量。--min_orthogroup_size:orthogroup比对的最小序列数量。--rooting_order:list文件,包含与分类群 (包括 scaffold taxa) 中物种序列标识符匹配的字符串片段列表,用于确定定根树中orthogroups的最基部类群。--bootstrap_replicates:rapid bootstrap分析的重复次数,需要--tree_inference raxml,默认100。--num_threads:用于RAxML的线程,默认1。debug
- 结果目录是空目录,没有生成结果文件
- 可能是序列中有中间终止密码子。在log文件中会出现。
- 查看序列中是否有.符号,要么删除有中间终止密码子的序列,要么把中间终止密码子符号(.)换成gap符号(-):
sed "s/\./-/g" old.fa >new.fa。
- 结果目录是空目录
- log文件中出现
Skipping sequence MLP_m64284e_210910_055234/158926758/ccs_MP021065 in orthogroup 3510 not in codon frame。cds序列和pep序列不一致,这个提示代表pep序列有但cds序列没有。 - 删除
7.8. KaKsAnalysis模块
7.8.1. 运行
- 进行旁系同源分析
PlantTribes/pipelines/KaKsAnalysis --coding_sequences_species_1 PlantTribes/test/species1.fna --proteins_species_1 PlantTribes/test/species1.faa --comparison paralogs --min_ks 0.02 --max_ks 4.0 --num_threads 10- 输出文件:
- kaksAnalysis_dir/species1.fna - species1 input coding sequences (CDS)
- kaksAnalysis_dir/species1.faa - species1 input amino acids (proteins)
- kaksAnalysis_dir/species1.fna.blastn.paralogs - species1 self blastn results
- kaksAnalysis_dir/species1.fna.blastn.paralogs.rbhb - species1 paralogous pairs
- kaksAnalysis_dir/species1.fna.blastn.paralogs.rbhb.kaks - species1 ka/ks analysis results
- 进行直系同源分析
PlantTribes/pipelines/KaKsAnalysis --coding_sequences_species_1 PlantTribes/test/species1.fna --proteins_species_1 PlantTribes/test/species1.faa --comparison orthologs --coding_sequences_species_2 PlantTribes/test/species2.fna --proteins_species_2 PlantTribes/test/species2.faa --min_ks 0.02 --max_ks 4.0 --num_threads 10- 输出文件:
- kaksAnalysis_dir/species1.fna - species1 input coding sequences (CDS)
- kaksAnalysis_dir/species1.faa - species1 input amino acids (proteins)
- kaksAnalysis_dir/species2.fna - species2 input coding sequences (CDS)
- kaksAnalysis_dir/species2.faa - species2 input amino acids (proteins)
- kaksAnalysis_dir/species1.fna.blastn.orthologs - species1 vs species2.blastdb blastn results
- kaksAnalysis_dir/species2.fna.blastn.orthologs - species2 vs species1.blastdb blastn results
- kaksAnalysis_dir/species1and2.fna.blastn.orthologs.rbhb - species1 vs species2 orthologous pairs
- kaksAnalysis_dir/species1and2.fna.blastn.orthologs.rbhb.kaks - species1 vs species2 ka/ks analysis results
- 进行旁系同源分析,拟合至多4个多元正态成分的混合模型,以确定基因组中显著的重复事件
PlantTribes/pipelines/KaKsAnalysis --coding_sequences_species_1 PlantTribes/test/species1.fna --proteins_species_1 PlantTribes/test/species1.faa --comparison paralogs --fit_components --num_of_components 4 --min_ks 0.02 --max_ks 4.0 --num_threads 10- 输出文件:
- kaksAnalysis_dir/species1.fna - species1 input coding sequences (CDS)
- kaksAnalysis_dir/species1.faa - species1 input amino acids (proteins)
- kaksAnalysis_dir/species1.fna.blastn.paralogs - species1 self blastn results
- kaksAnalysis_dir/species1.fna.blastn.paralogs.rbhb - species1 paralogous pairs
- kaksAnalysis_dir/species1.fna.blastn.paralogs.rbhb.kaks - species1 ka/ks analysis results
- kaksAnalysis_dir/species1.fna.blastn.paralogs.rbhb.kaks.components - significant components in the ks distribution of species1
- 进行直系同源分析,拟合至多4个多元正态成分的混合模型,以确定基因组中显著的重复事件
PlantTribes/pipelines/KaKsAnalysis --coding_sequences_species_1 PlantTribes/test/species1.fna --proteins_species_1 PlantTribes/test/species1.faa --comparison orthologs --coding_sequences_species_2 PlantTribes/test/species2.fna --proteins_species_2 PlantTribes/test/species2.faa --fit_components --num_of_components 4 --min_ks 0.02 --max_ks 4.0 --num_threads 10- 输出文件:
- kaksAnalysis_dir/species1.fna - species1 input coding sequences (CDS)
- kaksAnalysis_dir/species1.faa - species1 input amino acids (proteins)
- kaksAnalysis_dir/species2.fna - species2 input coding sequences (CDS)
- kaksAnalysis_dir/species2.faa - species2 input amino acids (proteins)
- kaksAnalysis_dir/species1.fna.blastn.orthologs - species1 vs species2.blastdb blastn results
- kaksAnalysis_dir/species2.fna.blastn.orthologs - species2 vs species1.blastdb blastn results
- kaksAnalysis_dir/species1and2.fna.blastn.orthologs.rbhb - species1 vs species2 orthologous pairs
- kaksAnalysis_dir/species1and2.fna.blastn.orthologs.rbhb.kaks - species1 vs species2 ka/ks analysis results
- kaksAnalysis_dir/species1and2.fna.blastn.orthologs.rbhb.kaks.components - significant components in the ks distribution of species1 vs species2 analysis results
7.8.2. 参数
- 必需参数
--coding_sequences_species_1:第一个物种的CDS fasta序列文件。--proteins_species_1:第一个物种的蛋白fasta序列文件。--comparison:成对序列比较以决定homolgous pairs。如果物种内比较:paralogs;如果物种间比较:orthologs(需要第二个物种数据)。
- 其他参数
--coding_sequences_species_2:当--comparison orthologs时,第二个物种的CDS fasta序列文件。--proteins_species_2:当--comparison orthologs时,第二个物种的蛋白fasta序列文件。--crb_blast:当--comparison orthologs时,使用条件reciprocal best BLAST来确定跨物种orthologs,而不是默认的reciprocal best BLAST。--min_coverage:同源对之间的成对序列覆盖长度的最小值。--recalibration_rate:重新校准ks。-主要适用于paralogous ks分析--codeml_ctl_file:PAML’s codeml control file用了执蛋白编码序列的ML分析--fit_components:拟合一个多正态组分的混合模型,用于确认ks分布。--num_of_components:多正态组分的数量。需要--fit_components。--min_ks:ks的下限。--max_ks:ks的上限。--num_threads:线程,默认1。
8. references
- tutorial:https://github.com/dePamphilis/PlantTribes/blob/master/docs/Tutorial.md
- github:https://github.com/dePamphilis/PlantTribes
- cite paper:https://www.frontiersin.org/journals/plant-science/articles/10.3389/fpls.2022.1011199/full#h3
- paper:https://academic.oup.com/gbe/article/15/1/evac183/6965378#394016961
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。


