1. Hifiasm软件的基本算法
Hifiasm软件由哈佛大学李恒团队在2021年2月份发表在Nature Methods上。
Hifiasm组装主要分为三步:
- 校正测序错误
- 构建分型字符串图(phased string graph)
- 单倍体分型组装
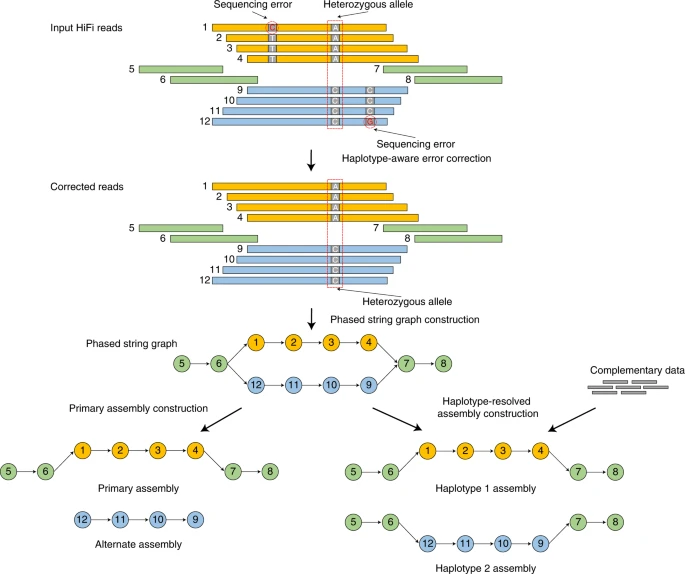
Figure 1. Hifiasm算法概况
1.1. 校正测序错误
- 尽管Hifi reads使用CCS测序模式已经进行了一轮校正,准确性已经比CLR测序模式高很多,但仍然会有部分测序(<1%)错误。
- Hifiasm进行所有序列的相互比对(all-versus-all)来校正可能的测序错误。
- 在比对中基于reads间的overlap关系来校正错误。如果在比对的同一个位置出现两种碱基类型(不考虑gaps),且每个碱基类型至少有3条reads支持,那么这个位置会被当作杂合位点(SNP)被保留。在这一步,Hifiasm可以对杂合SNP进行定相/分型(phasing)。
- 如果达不到上述条件的两碱基比对,两种碱基中较少的一种被视作测序错误,将被校正(默认三轮校正)。值得注意的是,Hifiasm只使用相同单倍型的数据进行纠错,从而避免过度校正,保留来自不同单倍型的杂合变异信息。
1.2. 构建分型字符串图(phased string graph)
- 基于第一步校正后的reads和reads之间的重叠关系(overlap),构建分型字符串图(phased string graph)。
- 以调整朝向的reads作为顶点(vertex),一致的overlap重叠区域作为边(edge),通过气泡 (bubble)的形式形成多条路径来表示杂合位点信息,因而可以保留下来基因组上全部的单倍型信息,以便后续对于单倍型的处理。
1.3. 单倍体分型组装
HiFi-only assembly 模式:如果没有其他数据,如亲本数据,Hi-C数据,使用此模式进行单倍体分型组装。
- hifiasm 会随机选择每个气泡的一边构建primary assembly(与 Falcon-Unzip 和 HiCanu 类似的主装配)。
- 对于杂合基因组,由于存在一个以上的纯合haplotype,因此primary assembly可能还会包含haplotigs。
- HiCanu 依靠第三方工具(如 purge_dups20)来去除多余的haplotigs。Hifiasm 集成实现了 purge_dups 算法的一个变体,简化了组装流程。
2. Hifiasm(trio)算法:Trio-binning模式
如果样本的亲本也进行了测序,则可以使用Trio-binning模式。Trio-binning模式主要在单倍体分型组装步骤上与上面不同。
Hifiasm的trio-binning模式基于2018年发表的TrioCanu软件的trio-binning策略,并进行改进。
2.1. 早期软件TrioCanu
- 2018年开发的TrioCanu软件的trio-binning策略先将三代reads区分为来自父本、母本以及部分无法区分的reads,对区分后的reads分别组装获得了子代的两套单倍体序列。
- TrioCanu软件的trio-binning策略的一个主要缺陷是,一部分杂合子reads无法明确地划分为亲本单倍型:如果双亲在某个位点上都是杂合,那么这个位点无法给reads提供有效的kmer信息,并且reads不能被唯一地被分配给一个亲本单倍型;如果父本在一个位点是杂合的,而母本是纯合的,则reads也无法唯一地被分配给母本单倍型。在标准的trio-binning策略中,无法区分的杂合reads在两个亲本数据集中都会使用。因此,两个亲本的等位基因可能存在于一个单倍型组合中,从而导致错误的重复。另外还可能存在将reads错误划分到其中一个亲本的情况。
- 总言之,TrioCanu软件的标准trio-binning策略无法清楚地分离两个亲本单倍型。
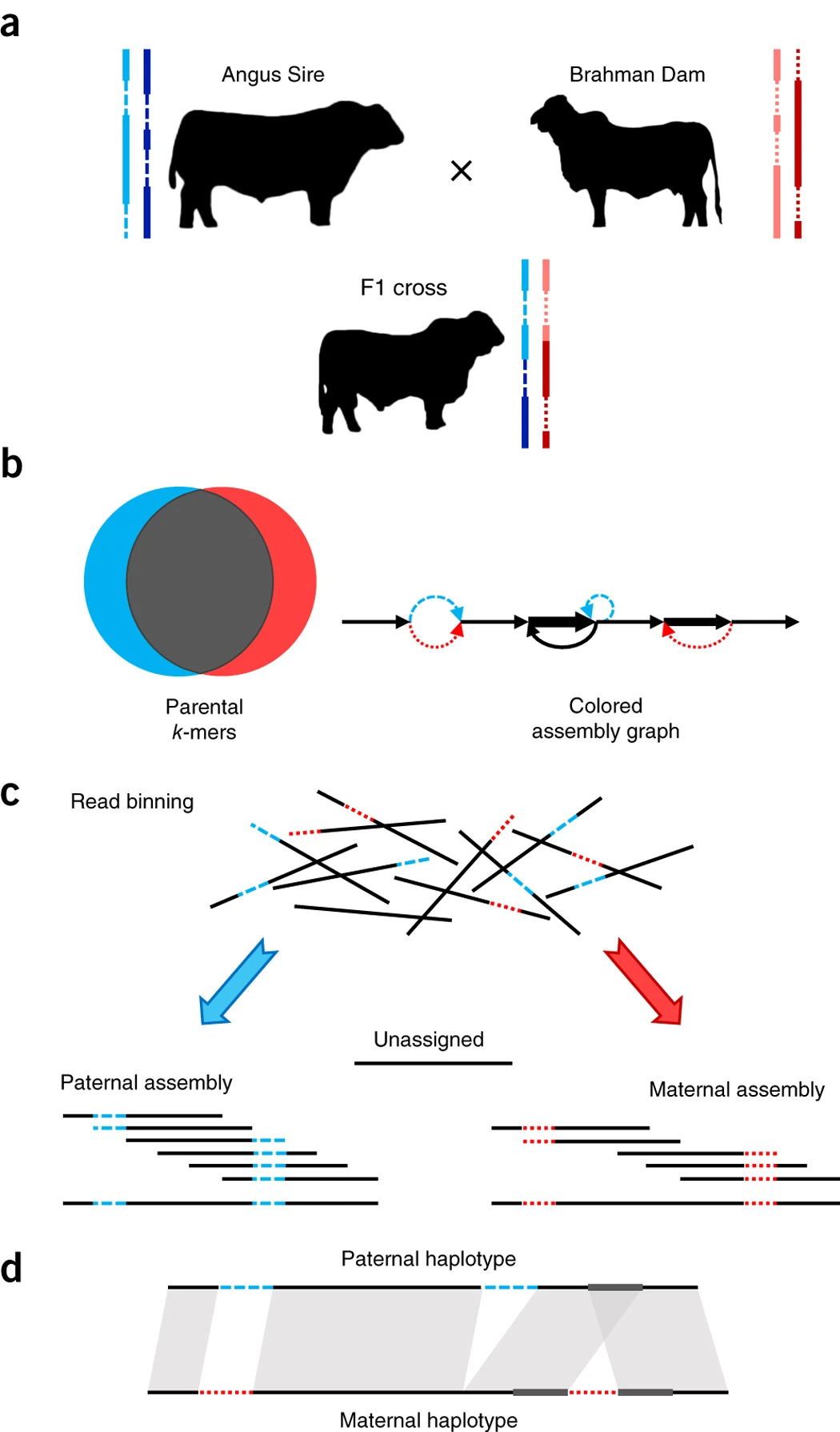
Figure 2. TrioCanu软件的trio-binning策略
2.2. Hifiasm的trio-binning模式
- 与TrioCanu软件的trio-binning策略不同,Hifiasm使用了graph-binning的策略对此进行了改进。
- Hifiasm 不预先划分reads,而是利用亲本特有的 k-mer trio binning 对字符串图(string graph)的reads进行标记。
- 因此在一个代表一对杂合等位基因的长bubble中,即使只有一小部分reads被正确标记,hifiasm也可以正确地将其分型。
- 标记之后,hifiasm 会有效地剔除母本的unitigs来生成父本的序列,反之亦然。
- Hifiasm的trio-binning模式的优势
- 通过这种方式,可以避免因为reads分型错误而引入的错误位点和组装断裂,避免错误地将双亲等位基因放在一个单倍型组装中,从而获得更完整和更准确的单倍体组装结果。
- 这种基于三元组分选的图(graph-based trio binning)可能会经过三元组(trio)中所有三个样本的杂合区域,对错误分型的reads的鲁棒性更高。
- Hifiasm的trio-binning模式修复分型错误
- 基于 Hi-C 或 Strand-seq 的分型可以明确地对大多数杂合reads进行分型,自然不会出现错误的重复。
- 不过,它们也有标准trio-binning策略的共同问题:分配给错误亲本单倍型的reads可能会破坏contigs(Figure 3)。
- 通过考虑 HiFi reads 分型和组装图(assembly graph)的结构,Hifiasm 可以识别并修复这类分型错误。
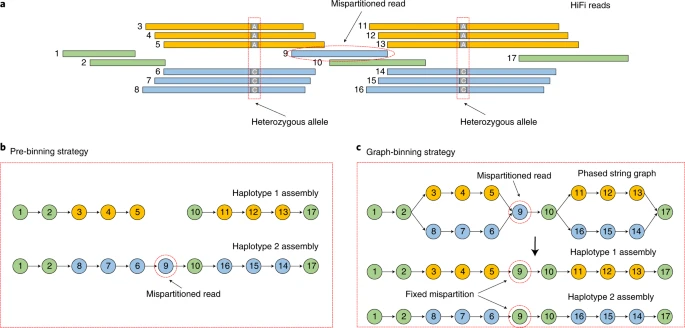
Figure 3. 错误的reads分型在Hifiasm的trio-binning模式中会被修正
3. 新算法Hifiasm(Hi-C):Hi-C Integrated assembly 模式
李恒团队2022年在Nature biotechnology上发表论文Haplotype-resolved assembly of diploid genomes without parental data(https://www.nature.com/articles/s41587-022-01261-x),在Hifiasm中引入了新算法Hifiasm(Hi-C),可以使用Hi-C Integrated assembly 模式进行单倍体分型组装。
3.1. Hi-C Integrated assembly模式
- Hi-C Integrated assembly模式针对PacBio HiFi (High-Fidelity) 长读长测序技术和Hi-C (High-Throughput Chromatin Confirmation Capture) 测序技术进行了全新的设计。
- 在无亲本数据的情况下,利用至少30倍覆盖度的HiFi数据和至少30倍覆盖度的Hi-C数据也可以获得二倍体生物的单倍型解决的组装结果。
- 它建立在分型 hifiasm 组装图 (assembly graphs)的基础上,但在序列分类(sequence partition)方面与已发表的 hifiasm (trio) 算法不同。
- 在 hifiasm graph中,每个节点(node)都是由相位正确的 HiFi reads组装而成的unitig,每条边(edge)代表两个unitigs之间的重叠。
- Hifiasm(trio)算法在亲本 k-mers 的unitigs中标记reads,但 Hifiasm(Hi-C)用 Hi-C reads对相对较短的unitigs进行分类。
3.2. Hifiasm(Hi-C)算法
- Hifiasm(Hi-C)算法,先检索unitigs中的 31-mers,并将 Hi-C reads 映射到这些unitigs中,而不进行详细的碱基比对。如果来自 Hi-C 片段的一对reads与两个unitigs上的两个远距离杂合子相匹配,就会在unitigs之间添加一个单倍型特异性 “link”,从而提供远距离的相位信息。
- 然后,把unitigs分类到两个类别中,使每个类别中的unitigs几乎没有冗余,并且每个类别共享许多 Hi-C “link”。
- 这样就将单元双分类问题简化为图最大切割(Max-Cut)问题,并通过随机算法(stochastic algorithm)找到接近最优的解决方案。
- 还考虑了组装图 (assembly graphs)的拓扑结构,以减少局部最优的概率。
- 最后,使用与 Hifiasm(trio)算法相同的图分选策略(graph binning strategy),生成最终的 Hifiasm(Hi-C)组装结果。
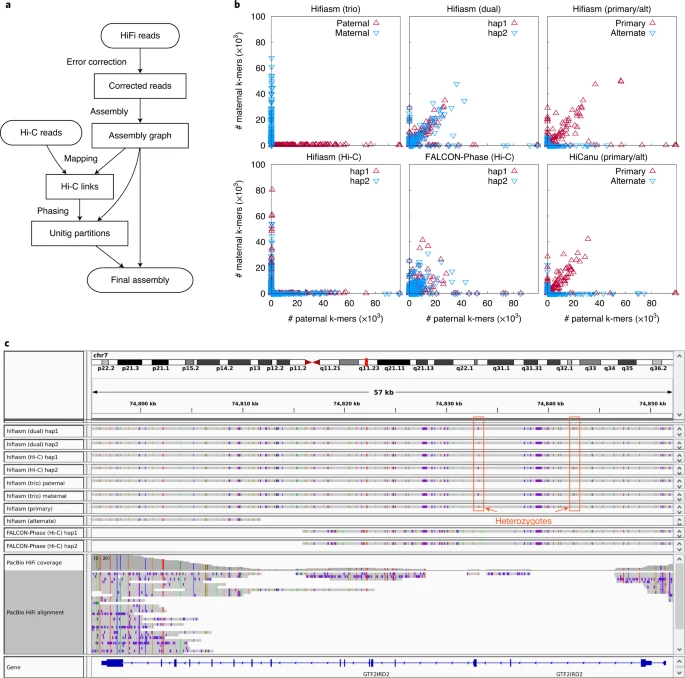
Figure 4. Hifiasm(Hi-C)算法
3.3. Hifiasm(Hi-C)算法与其他工具相比的优势
与现有基于Hi-C组装单倍体基因组的方法不同,Hifiasm(Hi-C)算法直接在 HiFi 组装图上运行,并将 Hi-C read mapping、分型(phasing)和组装紧密集成到一个单一的可执行程序中,而不依赖外部工具。它更易于使用,运行速度更快。
4. references
- Hifiasm paper:https://www.nature.com/articles/s41592-020-01056-5
- Hifiasm (Hi-C Integrated assembly 模式)paper:https://www.nature.com/articles/s41587-022-01261-x
- Hifiasm介绍:https://www.bilibili.com/read/cv18775152/
- https://www.jianshu.com/p/9dc0b5c5af81
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。


