1. 基因组测序
- 基因组测序简介
- DNA 测序是确定核酸序列(DNA 中核苷酸的顺序)的过程。它包括用于确定腺嘌呤、鸟嘌呤、胞嘧啶和胸腺嘧啶(A G C T)四种碱基顺序的任何方法或技术。
- 确定一个个体或一个细胞内的所有DNA序列的过程及基因组测序,通常是指细胞核基因组。
- 基因组测序技术
- 目前测序技术包括一代测序(Sanger测序)、二代测序(Illumina高通量测序)、三代测序(PacBio SMRT和nanopore测序)。

Figure 1. 测序技术的历史
2. 一代测序
1977年第一代DNA测序技术(Sanger测序)被Sanger团队首次开发,1986年被首次商业化。
2.1. Sanger测序原理
- 最常用的是链终止法(Chain-termination methods)进行Sanger测序。
- Sanger测序(链终止法)的原理是使用荧光或放射性标记的二脱氧核苷酸三磷酸酯(ddNTPs)和普通脱氧核苷酸三磷酸酯(dNTPs)共同作为原材料根据单链DNA模板在DNA引物和DNA聚合酶作用下在体外进行DNA链的延伸。由于ddNTPs缺乏在两个核苷酸之间形成磷酸二酯键所需的3-OH基团,所以ddNTPs会在结合后终止DNA链的延伸。
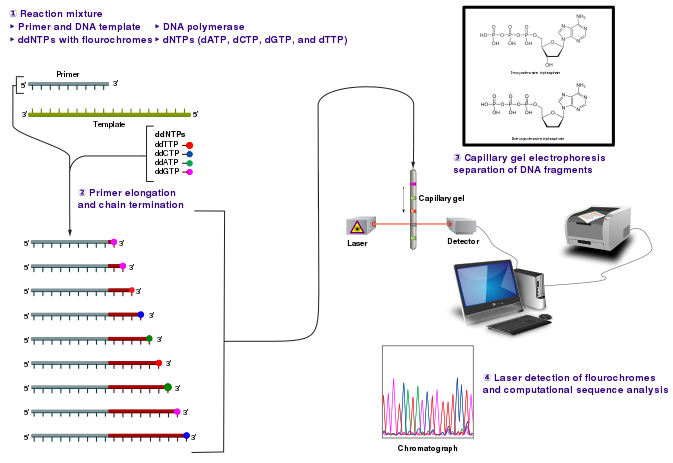
Figure 2. Sanger测序流程
- DNA 样品被分成四个独立的测序反应,每个反应体现都包含所有四种标准脱氧核苷酸(dATP、dGTP、dCTP 和 dTTP)和 DNA 聚合酶。每个反应只加入四种二脱氧核苷酸(ddATP、ddGTP、ddCTP 或 ddTTP)中的一种。脱氧核苷酸的浓度应比相应的双脱氧核苷酸的浓度高约 100 倍(例如 0.5mM dTTP : 0.005mM ddTTP),这样才能在转录完整序列的同时产生足够的片段(但 ddNTP 的浓度也取决于所需的序列长度)。
- 在模板 DNA 与结合引物进行多轮延伸后,将产生的 DNA 片段加热变性,并使用凝胶电泳按大小进行分离。
- 通常采用变性聚丙烯酰胺-尿素凝胶进行检测,四个反应分别在四条泳道(A、T、G、C泳道)中的一条进行。然后用自动射线照相法或紫外线照相法观察 DNA 条带,并可直接从 X 射线胶片或凝胶图像上读取 DNA 序列。

Figure 3. Sanger测序凝胶成像
2.2. Sanger测序特点和应用
- 优点:Sanger测序准确率约99.99%,是准确检测单核苷酸变异(SNP)和小插入/缺失(INDEL)的黄金标准方法。
- 缺点:一次只能对一条相对较短(300-1000 bp)的 DNA 片段进行测序。超过1000 bp的DNA片段难以分离,无法分辨凝胶成像中碱基顺序。
- 缺点:由于引物结合,序列前 15-40 个碱基的质量较差,以及 700-900 个碱基后测序痕迹的质量下降。
- 应用:Sanger测序被广泛用于检测已知的家族变异、验证通过 NGS 获得的结果以及某些单基因测序。
3. 二代测序
第二代测序技术也叫下一代测序技术(next generation sequencing, NGS)在1993-1998年间出现,2005年开始投入商业使用。
最常用的NGS测序是在Illumina HiSeq平台上进行边合成边测序(Sequencing by synthesis,SBS),通常是双端测序(Paired end),准确率约99.9%。
3.1. 二代测序(边合成边测序,Sequencing by synthesis,SBS)的步骤
- 体外PCR扩增,增强信号,制备DNA测序文库。
- 将待测序的DNA附着在固体载体上,在固体载体上生成单链DNA。
- 使用DNA聚合酶、dNTPs等以固体载体上的单链DNA为模板进行DNA合成。
- 对空间隔离的DNA模板同时进行大规模并行的边合成边测序。
3.2. 二代测序特点和应用
- 优点:高通量:Illumina一次运行可测序1M-43G条reads(每条reads 50-400个碱基),可以对整个基因组进行测序。
- 优点:价格低:相对第一代测序技术,在高通量的情况下价格比第一代低很多。
- 准确率(99.9%)低于第一代测序技术(99.99%),有时需要第一代测序技术的验证。
- 缺点:读长(reads)短:插入序列长度常为150bp,很少超过1000bp。
- 测序速度:Illumina一次运行需要1-11天。
4. 三代测序
第三代测序技术又叫长读长测序(Long-read sequencing),常用的测序技术包括Pacific Biosciences(PacBio)公司的单分子实时测序技术(Single-Molecule Real Time Sequencing technology, SMRT)和 Oxford Nanopore Technology公司的Nanopore的纳米孔测序技术。2008年以来一直在发展中。
4.1. 三代测序的特点
- 优点:读长(reads)长:SMRT测序reads的N50约为30kbp,最长reads可超过100kbp。
- 缺点:准确率低:SMRT测序CLR模式获得的reads的准确率约为87%,但2019年推出的CCS模式经过一致性校正后准确率可达99.9%。
- 中等通量:SMRT测序每次运行生成4M条reads。
- 便携性和速度:SMRT平台一次运行需要0.5-20小时。Oxford Nanopore Technology开发的MinlON测序仪,它只有U盘大小,可以便携和快速的检测。
- 可以直接检测表观遗传标志物:
- 不同于二代测序的碱基质量标准Q20/Q30,三代测序由于其随机分布的碱基错误率,其单碱基的准确性不能直接用于衡量数据质量。
4.2. 三代测序的应用
- 基因组组装:长读长组装起来更快更准确。
- 识别结构变异:短读长无法识别的结构变异用长读长的reads可以识别了。
4.3. PacBio SMRT测序
Pacific Biosciences(PacBio)公司三代测序平台Sequel & Sequel II基于零模波导特性(zero-mode wave-guides, ZMWs)的单分子实时测序技术(SMRT)进行测序。
4.3.1. PacBio三代建库
- PacBio三代建库是在已打断为一定长度(10-20kb或更长)的双链DNA分子两端连接带有发卡(hairpin)结构的PacBio接头(adaptor),使DNA分子形成“哑铃形”SMRTbell文库。
4.3.2. SMRT测序原理
- SMRT 测序是以SMRT Cell为载体,SMRT Cell是纳米制造的,不可回收的消耗品,也叫芯片。每个SMRT Cell上布满了数百万个零模波导孔(ZMW),也被称为well。零模波导孔(ZMW)是单分子实时测序的最小场所。Sequel 系统一个SMRT Cell上面 1M(一百万)个ZMWs, Sequel II系统一个Cell上支持8M个ZMWs。
- 测序时DNA聚合酶和一条模板分子被瞄定在ZMW孔底部进行反应,位于小孔底部的激发光能够激发核苷酸底物上的荧光标记,进而通过监测系统的相机(CCD)将荧光信号记录下来,从而获得碱基信息。这一过程在环化的文库上不断重复进行,从而完成测序。
- 整个测序过程 DNA 分子不需要经过PCR扩增,实现了对每一条DNA分子的单独测序。
4.3.3. SMRT测序模式
PacBio sequel II平台支持CLR(Continuous Long Reads)和CCS(Circular Consensus Sequencing)两种测序模式。
- CLR模式适用超长片段文库(> 25 kb),存储有效数据的文件一般命名为* .subreads.bam。对subreads不再进行后续处理。
- CCS模式则适用于普通长度片段文库(< 25 kb),存储数据的* .subreads.bam文件(subreads)需要后续处理。由于测序过程中每条polymerase read中包含同一分子的多条pass信息,通过一致性校正后(intra-molecular consensus),得到一条唯一的read,称为CCS read,这个校正过程会显著提升测序准确率(从86%提升到99.9%)。质量值大于Q20的CCS read称为HiFi reads(High fidelity reads),存放于.hifi_reads.bam文件中,这个文件将用于下游的信息分析。
4.4. Nanopore 纳米孔测序
相较PacBio的三代测序技术,Oxford Nanopore Technology公司的Nanopore测序(又叫纳米孔测序)技术使用的较少。
- Nanopore测序原理
- Nanopore测序技术采用的是一种与SMRT不同的原理,即单链 DNA 或 RNA 分子穿过纳米孔,记录核苷酸通过孔时的周围电场的电流变化。
- Nanopore测序平台
- Nanopore测序的特点是便携性和多功能性。手持式 MinION 设备使研究人员能够在传统实验室之外进行测序实验,从而实现对传染病爆发、环境研究甚至太空任务的实时监控。
- PromethION 是该技术的高通量版本,可提升测序能力以应对更大的项目,包括全基因组测序和元基因组学。
- Nanopore测序特点
- 缺点:插入片段长度最高可达2.3Mbp,通常比PacBio三代reads(30kb)短。
- 测序reads的准确率约为92-97%。
- 优点:便携和快速。
- 优点:纳米孔测序的实时分析。
5. references
- 维基-DNA sequencing:https://en.wikipedia.org/wiki/DNA_sequencing#:~:text=Sequencing%20is%20used%20in%20molecular,and%20identify%20potential%20drug%20targets.
- 维基-Sanger测序:https://en.wikipedia.org/wiki/Sanger_sequencing
- https://biopic.pku.edu.cn/gtlcxzx/ptjs/swxxfx/index.htm
- https://www.cnblogs.com/leezx/p/6136297.html
- 第一代到第三代基因组测序技术原理:https://www.cnblogs.com/huangshujia/p/3233693.html
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。


