1. 进化树
进化树的相关知识可以参考博文进化树相关知识。
2. 进化树的绘制
进化树的绘制是指把newick格式的树文件转换成树图形,有许多软件可以使用,在线网页版(itol,EvolView),也有R包(ggtree),或者软件figtree。
如果是简单的树绘制,推荐在线网页itol;如果需要精细的注释,推荐R包ggtree。
2.1. 在线绘制进化树
2.2. R包ggtree【推荐】
R包treeio用于解析各种格式的进化树文件,ggtree用于绘制进化树和注释。
2.2.1. 树文件的导入
2.2.1.1. treeio导入树
treeio支持标准格式树(包括Newick树格式,NEXUS树格式,New Hampshire eXtended(NHX)树格式,Jplace树格式)和建树软件输出树(RAxML,iqtree,BEAST,MrBayes,PAML(BASEML,CODEML),r8s,MEGA,HyPhy,Phylip,以及生成Jplace格式树的EPA和PPLACER),可以用文本输入和文件输入两种形式导入和解析树。
treeio tutorial解释了各种建树软件输出的树包含的信息。
- 文本输入
当树文件的文本比较简单时,可以直接把文本读到变量中,再解析。
Newick格式树
read.newick():用read.newick读取Newick格式的树文本,有枝长信息,缺点是没有办法添加注释信息1
2
3library(treeio)
treetext<-"((((Mc:0.418672,Eg:0.201388):0.148853,((Pt:0.219587,(Cs:0.209405,(Gr:0.224188,At:0.492461):0.027774):0.012564):0.019343,(Pp:0.194515,(Mt:0.354580,Cl:0.338644:0.022063):0.019111):0.011710):0.029914,Vv:0.169100):0.241611,Mg:0.241611);"
tree <- read.newick(textConnection(treetext))NHX格式树
read.nhx():用read.nhx()读取NHX格式的树文本,节点后中括号内[&&NHX]添加值注释信息,用冒号:分隔多个注释信息,缺点是没有办法添加范围注释,比如95%置信区间注释1
2treetext<"((((Mc:0.418672[&&NHX:I=5728:D=1170:G=Malvids],Eg:0.201388[&&NHX:I=1420:D=2519:G=Malvids])[&&NHX:C=1]:0.148853,((Pt:0.219587[&&NHX:I=5355:D=1003:G=Fabids],(Cs:0.209405[&&NHX:I=1026:D=3516:G=Malvids],(Gr:0.224188[&&NHX:I=4875:D=998:G=Malvids],At:0.492461[&&NHX:I=1776:D=2891:G=Malvids]):0.027774[&&NHX:C=2]):0.012564[&&NHX:C=3]):0.019343[&&NHX:C=4],(Pp:0.194515[&&NHX:I=1159:D=2949:G=Fabids],(Mt:0.354580[&&NHX:I=3310:D=1984:G=Fabids],Cl:0.338644[&&NHX:I=794:D=3735:G=Fabids]):0.022063[&&NHX:C=5]):0.019111[&&NHX:C=6]):0.011710[&&NHX:C=7]):0.029914[&&NHX:C=8],Vv:0.169100[&&NHX:I=1522:D=3045:G=Vitales]):0.241611[&&NHX:C=9],Mg:0.241611[&&NHX:I=2278:D=1909:G=Asterids])[&&NHX:C=10];"
tree <- read.nhx(textConnection(treetext))
- treeio导入树文件
treeio支持解析大部分软件输出的树文件
| 解析函数Parser function | 功能 |
|---|---|
| read.newick | 解析Newick格式文件 |
| read.nexus | 解析NEXUS格式文件 |
| read.nhx | 解析NHX格式文件 |
| read.jplace | 解析jplace格式文件,包括EPA和pplacer的输出文件 |
| read.jtree | 解析jtree格式文件 |
| read.fasta | 解析fasta序列 |
| read.iqtree | 解析IQ-Tree的Newick格式输出,支持解析SH-aLRT和UFBoot支持率 |
| read.raxml | 解析RAxML的输出 |
| read.r8s | 解析r8s的输出 |
| read.mrbayes | 解析MrBayes的输出 |
| read.beast | 解析BEAST的输出 |
| read.astral | 解析ASTRAL的输出 |
| read.mega | 解析MEGA的Nexus格式输出 |
| read.codeml | 解析CodeML的输出,rst和mlc文件 |
| read.paml_rst | 解析rst文件,BaseML或CodeML的输出 |
| read.hyphy | 解析HyPhy输出文件 |
| … | … |
用对应的解析函数读取相应格式的文件,例如读取Newick格式文件用tree<-read.newick("newick.tre"),读取beast输出的树文件用tree<-read.beast("beast.tre")。
- read.mega/read.beast读取NEXUS格式文件
- NEXUS格式把Newick树和其他信息组合,每种信息单独成块(blocks),可扩展块来实现添加新类别的信息,NEXUS格式可以看作Newick树加注释信息的组合。
- NEXUS格式树包括TAXA块和TREE块,常常也会包括储存序列比对(Phylip格式)的DATA块,有些软件有自己特定的块。
- 每个块都是
Begin Blockname;开头,End;加一个空行结尾。 - NEXUS格式的优点是可以实现尽可能地注释树,包括值注释和区间注释(例如95%置信区间),缺点是格式稍微复杂,必须文件读取。
- 我自己尝试用read.nexus读取NEXUS文件,读取没问题,但如果想使用注释信息(比如geom_text/geom_label2使用注释,geom_range来显示区间注释)就会报错;但是用read.mega/read.beast读取NEXUS文件就可以顺利使用注释信息,查看了treeio里的示例文件,read.mega和read.beast里的NEXUS文件格式是一样的,区别在于beast只能读NEXUS文件,mega还可以都Newick文件。
一个NEXUS文件的例子,treeio包自带的mtCDNA_timetree.nex文件,可以参考示例文件格式自己写一个NEXUS格式的树文件用于ggtree:
1 | #NEXUS |
在代表样品的序号后用中括号[&]提供注释信息,多个注释用逗号分隔,如果提供的注释信息值是区间,用大括号包含区间。
2.2.2. ggtree画树
ggtree函数是ggplot()的扩展,ggtree()相当于ggplot() + geom_tree() + xlab(NA) + ylab(NA) + theme_tree()的简单组合。ggplot2可添加的图层都可以直接应用于ggtree。
2.2.2.1. 基础画树
1 | library(ggtree) |
2.2.2.2. 基础注释
基础注释函数用法
包含注释函数的基本参数用法1
2
3
4
5
6
7
8
9
10
11
12
13ggtree(tree) \ #画树
+ geom_tiplab() \ # 末端节点标上物种
+ geom_rootedge() \#添加根节点的枝长线,如果树文件中有根节点枝长则直接可使用
+ geom_point2() \ #节点上标记圆点
+ geom_text2(aes(label=node)) \ #节点上做文字标记。label指定内容,这里指定节点上标记节点号,可以用这个确定树图上对应的节点号
+ geom_label2(aes(label=node)) \#节点上做标签标记。label指定内容,这里指定节点上标记节点号。
+ geom_cladelabel(20,label="legumes",angle=90) \#用条带和文本注释一个clade(node20),label指定文本内容,angle指定文本角度
+ geom_strip(9,10,label="legumes") \#用条带指定node9到node10的位置,label指定文字内容
+ geom_hilight(node=20) \#用矩形高亮node20号分支,用法和geom_highlight()好像是一样的
+ geom_range("length_0.95_HPD") \#在节点上画长条形的分歧时间的95%置信范围,值来自树文件中的length_0.95_HPD注释值
+ geom_taxallink(taxa1=12, taxa2=20) \#用曲线连接node12和node20
+ geom_treescale() \#添加比例尺
+ xlim(NA,1) \#如果内容超出边界,就用xlim拓展显示x轴的范围常用注释参数
包含注释函数的常用参数用法1
2
3
4
5
6
7
8
9
10
11ggtree(tree, layout="circular", size=0.8, branch.length='none') \ #画树,layout指定布局类型,默认是rectangular;size指定枝的粗细;branch.length指定无枝长信息
+ geom_tiplab(color="black", size = 4, hjust = -0.1, align=T, linesize=0.7) \ # 末端节点标上物种,size文字大小,hjust是离进化枝线条的距离,align=T文字间左对齐,linesize设置虚线尺寸。
+ geom_rootedge(rootedge = 1,size=1) \#添加根节点的枝长线,如果树文件中有根节点枝长则直接可使用,否则可以用rootedge设置根节点枝长(不设置为0),size设置粗细。结合xlim来显示。
+ geom_point2(color="#6FE1F8", size=5, alpha=0.7) \ #所有节点上标记圆点,配色和透明度
+ geom_point2(aes(subset=(node==16)), color='darkgreen', size=5) \#在16号节点标记绿色圆点。其中subset是取子集,符合条件的才注释;color设置颜色,size设置大小。
+ geom_text2(aes(label=branch.length,hjust=-0.1),size=3, fontface = "bold") \#节点上标记枝长;size设置大小,fontface设置文本加粗显示
+ geom_text2(aes(subset=!isTip, label=node), hjust=-0.3, size=3, color="deepskyblue4") \ #非tip节点标记上node号
+ geom_label2(aes(x=0.79,label=D),color="black",vjust=-0.2,fill="#7FFF00",alpha=0.6) \#做标记,aes里的x指定位置,label指定内容
+ geom_strip(9,10,label="legumes",offset = 0.06,offset.text = 0.012,color = "#FF6100",barsize = 1.5,fontsize = 5,angle = 90,hjust = 0.5) \#用条带指定node9和node10之间的分支clade,label指定文字内容,offset指定条带和文字与末端节点的距离,offset.text指定文字与条带的距离,barsize指定条带尺寸,fontsize指定文字尺寸,hjust指定文字位置
+ geom_hilight(node=15,fill="firebrick",alpha=0.6) \#用矩形高亮一个分支
+ geom_range("length_0.95_HPD", color='red', size=2, alpha=.5, center='length') #在节点上画长条形的分歧时间的95%置信范围,值来自树文件中的length_0.95_HPD注释值,让中心点位于length注释值其他设置
1
2
3ggtree(tree)
+ scale_x_reverse() #反转x轴
+ coord_flip() #置换x轴和y轴
2.2.2.3. 改变树的结构
改变树的结构包括取子树(viewClade),坍塌(collapse),扩展(expand),旋转(rotate),分组(groupClade)等操作,下面两种函数用法是等价的:
- rotate(p,node=12)
- p %>% rotate(node=12)
当需要多次改变树的结构时,推荐第二种用法,比如p2 <- p1 %>% flip(37,48) %>% flip(3,4) %>% flip(13,14) %>% flip(25,26) %>% flip(28,29) %>% flip(55,56)即可依次改变。
显示子树
1
2p <- ggtree(tree)+geom_tiplab()
viewClade(p, MRCA(p, 10,15)) #显示10-15节点的子树部分旋转枝
1
2flip(p, 15, 17) #交换15号节点和17号节点代表的枝的位置,15和17号共享同一个祖先节点16。
rotate(p, 16) #在16号节点把上下两个枝镜像翻转180°,与flip的区别是rotate同时改变了15和17号节点内所有子节点的顺序坍塌和扩展枝
1
2p2 <- collapse(p, node=16) + geom_point2(aes(subset=(node==16)), shape=21, size=5, fill='green') #坍塌21号节点,用绿色圆点标记
expand(p2, node=21) #把p2坍塌的21号节点扩展开缩小和放大枝
1
2scaleClade(p, node=15, scale=0.1) #缩小15号节点及子节点的枝,压缩尺度为0.1,拓扑和枝长仍然保留,但枝间距缩小为原来的0.1倍。常用于tip比较多的系统树上缩小不重要的部分,但不坍塌。
scaleClade(p, node=17, scale=1.5) #放大17号节点及子节点的枝,放大尺度为1.5,枝间距放大为原来的1.5倍。常用于突出重要的枝。分组
单系分组用groupClade,多系/并系分组用groupOTU。1
2
3
4
5p3 <- groupClade(p, c(15,17)) #节点15及子节点分为一组,节点17及子节点分为一组,赋予group属性,适用单系分组。
ggtree(p3, aes(color=group)) + scale_color_manual(values=c("black", "red", "blue")) #用group属性注释枝的颜色,默认颜色为黑色,15节点代表的组为红色,17节点代表的组为蓝色
p4 <- groupOTU(p, focus=c("A","D","G")) #A,D,G分为一组,赋予group属性
ggtree(p4, aes(color=group)) #用group属性注释枝的颜色,A,D,G所在的枝和共享的枝一种颜色,其他枝另一种颜色。A,D,G可以是多系/并系。
2.2.2.4. 可视化数据并与系统发育树关联 —— facet_plot
可能有不同的数据类型,并希望将它们可视化并与树关联对齐,可用facet_plot函数来处理。
可视化图的类型:点图dotplot/直方图barplot/堆叠图stacked barplot/热图/箱线图boxplot。
ggtree中定义了操作符%<+%,来添加数据。添加之后,用户的数据对ggplot是可见的。可以用于树的注释。
- 可视化点图dotplot —— geom_point
1
2
3
4
5
6
7tr <- rtree(30) # 用treeio的rtree函数生成30个tip的随机树
p <- ggtree(tr)
d1 <- data.frame(id=tr$tip.label, location=sample(c("GZ", "HK", "CZ"), 30, replace=TRUE)) #从("GZ", "HK", "CZ")中随机生成30个采样地点location数据
p1<- p %<+% d1 + geom_tippoint(aes(color=location)) #给树的端点根据location着色
d2 <- data.frame(id=tr$tip.label, val=rnorm(30, sd=3)) #生成两列数据,分别是tip.label和随机生成的30个sd为3的数据值,均值mean默认为0
p2 <- facet_plot(p1, panel="dot", data=d2, geom=geom_point, aes(x=val), color='firebrick') + theme_tree2() #用ggtree的facet_plot的geom_point函数画点图
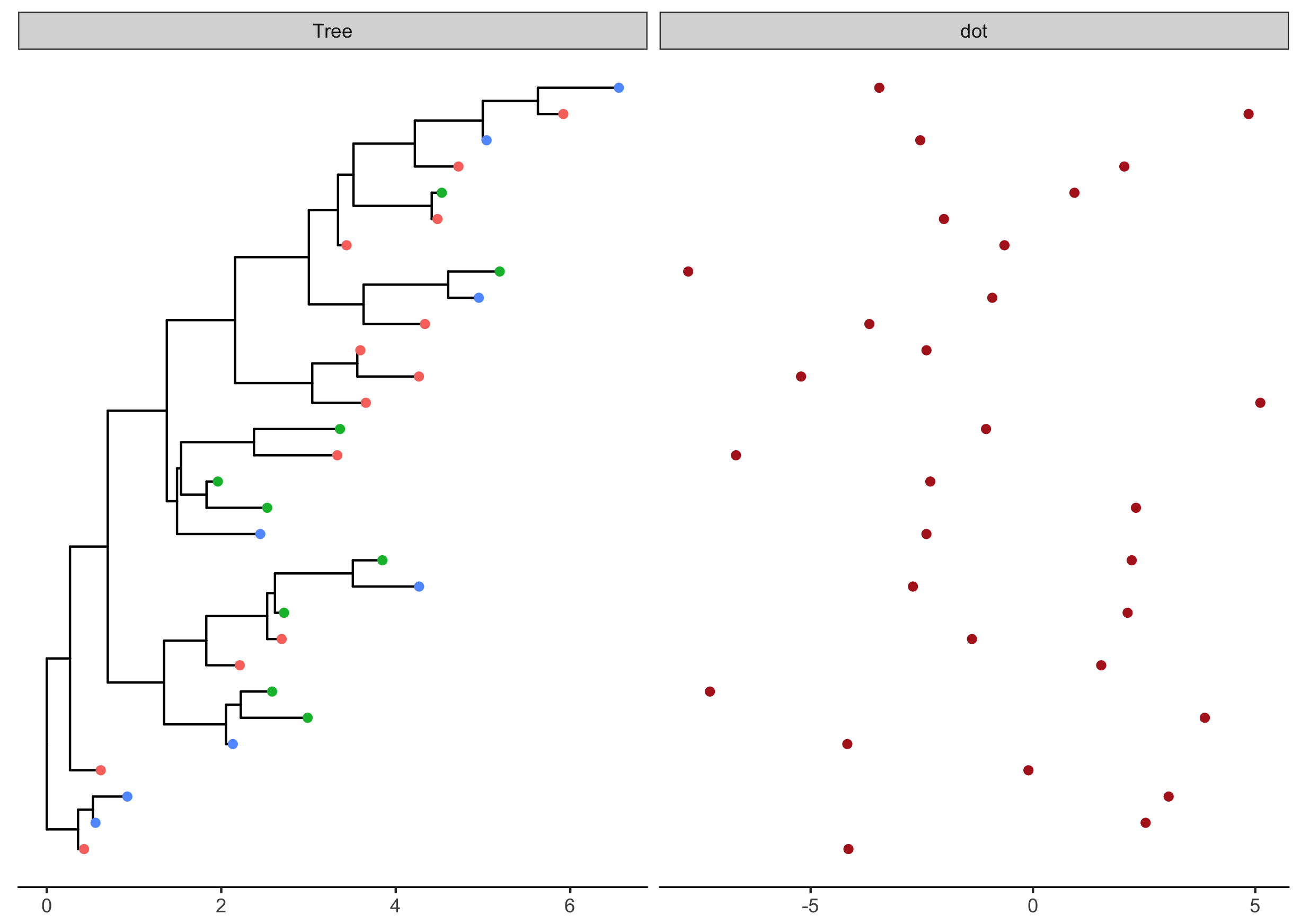
Figure 1. p2 dotplot
from guangchuangyu’s blog
- 可视化堆叠直方图stacked barplot —— geom_barh
大多ggplot2的geom绘制垂直版本的图形对象,通过ggstance包可以制作水平版本的数据图geom,方便画直方图/堆叠直方图/热图/箱线图与系统发育树对应。
包括geom_barh(),geom_histogramh(),geom_linerangeh(),geom_pointrangeh(),geom_errorbarh(),geom_crossbarh(),geom_boxploth(),geom_violinh()。1
2
3
4
5
6
7
8library(ggstance)
d3 <- data.frame(id = rep(tr$tip.label, each=2),
value = abs(rnorm(60, mean=100, sd=50)),
category = rep(LETTERS[1:2], 30)) #随机生成3列数据,分别是tip.label,随机值,代表category的A/B
p3 <- facet_plot(p2, panel = 'Stacked Barplot', data = d3,
geom = geom_barh, #用geom_barh画水平版本的堆叠直方图
mapping = aes(x = value, fill = as.factor(category)),
stat='identity' )
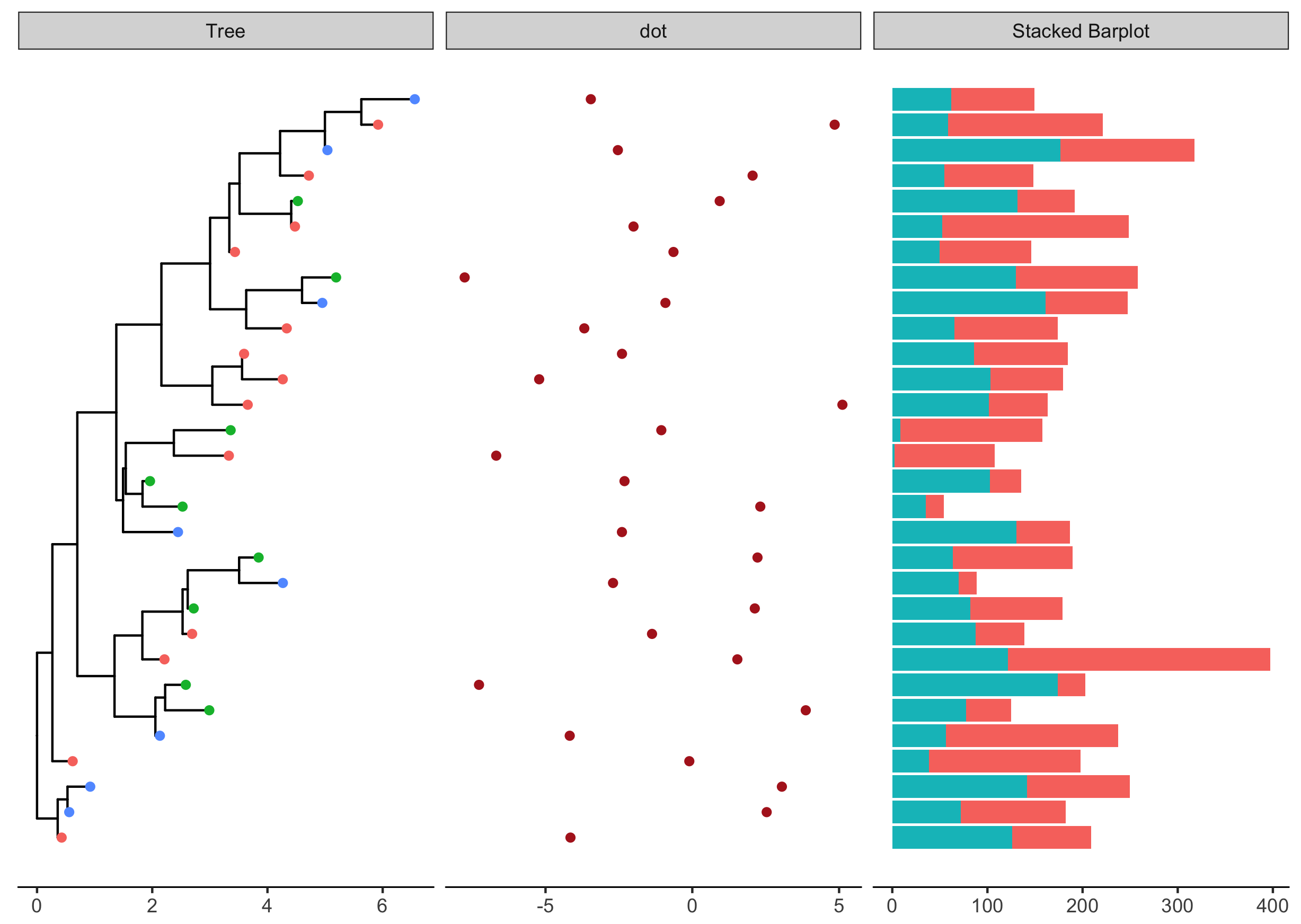
Figure 2. p3 barplot
from guangchuangyu blog
- 可视化箱线图boxplot —— geom_boxploth
1
2
3
4
5
6d4 = data.frame(id=rep(tr$tip.label, each=20),
val=as.vector(sapply(1:30, function(i)
rnorm(20, mean=i)))
)
p4 <- facet_plot(p3, panel="Boxplot", data=d4, geom_boxploth,
mapping = aes(x=val, group=label, color=location)) #用geom_boxploth画水平版本的箱线图
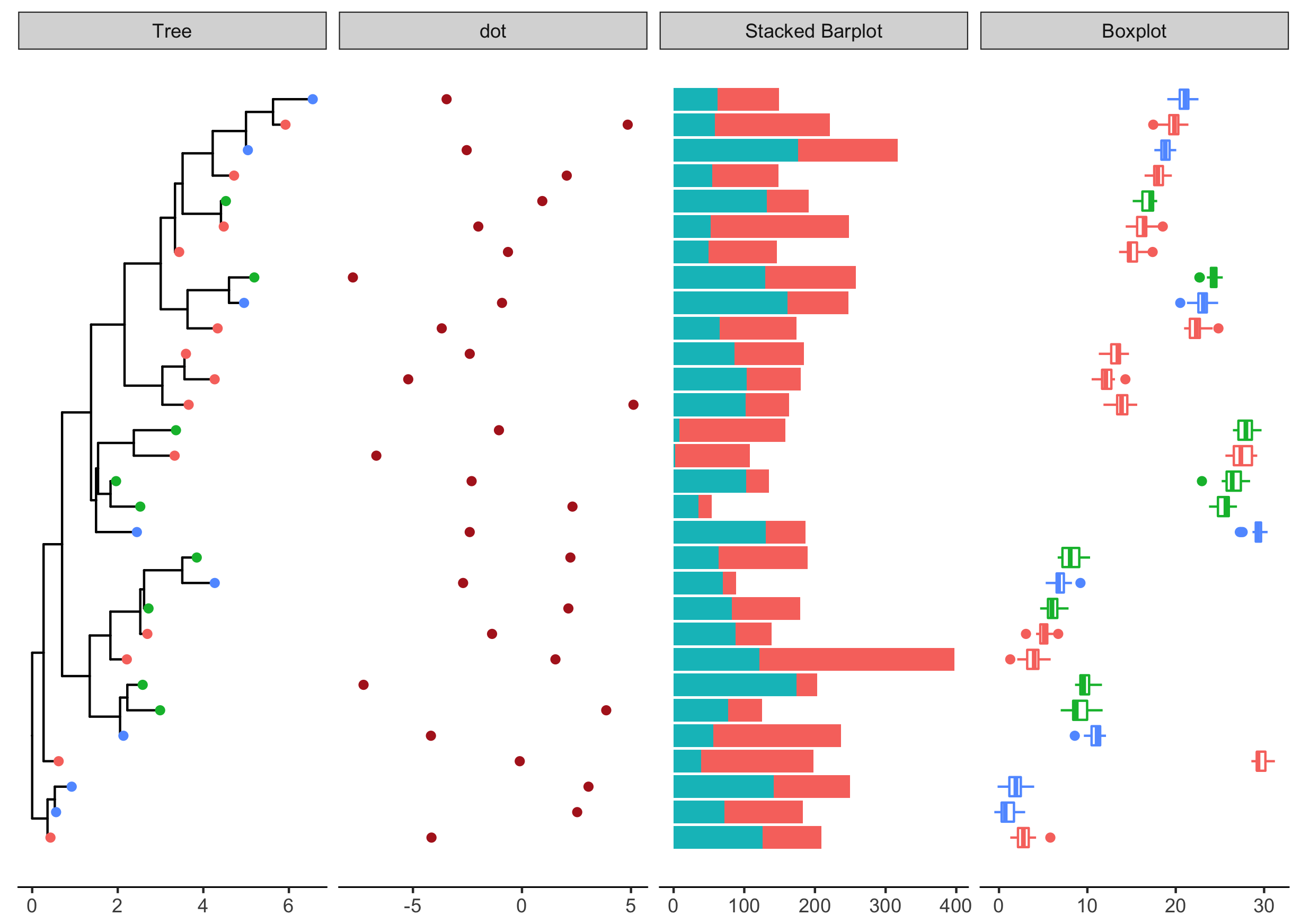
Figure 3. p4 boxplot
from guangchuangyu blog
2.2.2.5. 时序树(chronogram)
时序树上每个节点的数字代表的是分歧时间,端点一般对齐(因为端点的所有样本都对应当下的采样时间),画时序图需要先有时间标定的树文件,树上的枝长代表时长,一般会再添加时间尺度轴。
- 画时序树
- 通过
ggtree(tree, mrsd="2020-01-01")中的mrsd指定采样日期(即tip端点时间),mrsd代表most recent sampling date,指定采样时间 - 通过
+theme_tree2()来在树下添加一个时间尺度轴,默认是以年为单位 - 可用deeptime包向ggtree添加地质时间尺度
实现采样时间(mrsd)和内部节点时间来缩放树
- 读取时序树
读取时序树与读取任何系统发育树的方法一样,读取任何时间代表枝长的树文件即可。
- 可以用treeio解析估计时间的软件(例如beast,r8s)输出的时序树;
- 也可以把时间写入枝长位置,以文本的形式读取时序树;
- 时间尺度轴的变化
直接用
theme_tree2() +scale_x_continuous()来设置时间尺度轴的显示1
ggtree(tree) + theme_tree2() +scale_x_continuous(limits=c(-1,4),breaks = seq(0,4,1),labels = c(4,3,2,1,0),expand = c(0.1,0.1)) + geom_treescale(x=3,y=20,color="red") #画树,theme_tree2()在树下方加时间尺度轴,这里假设是[0,4],用scale_x_continuous()限制时间尺度轴的显示和刻度,limits=c(-1,4)设置尺度轴显示范围为-1到4(同样也是图的横向显示范围),breaks设置值的显示范围为0到4,每隔1显示一个值,labels设置显示的值,可以直接设定c(4,3,2,1,0)使时间尺度轴倒序显示,expand设置绘图左右范围的空白大小;geom_treescale()在右上角添加比例尺
用
revts设置时间尺度轴翻转1
2
3
4
5
6p <- ggtree(tree) + theme_tree2()
p1 <- revts(p) #翻转时间尺度轴,比如[0,4]翻转成[-4,0];注意使用revts后,xlim的范围也要相应调整。
library(ggplot2) #scale_x_continuous是ggplot2的函数
p2 <- p + scale_x_continuous(labels = abs) #把时间尺度轴的标签显示为绝对值abs的形式,scale_x_continuous是ggplot的函数
p3 <- revts(p2) #把p2时间尺度轴翻转,由于p2用了绝对值,效果是把[0,4]翻转成了[4,0]
p4 <- p1 + scale_x_continuous(breaks=c(-4:0), labels=abs(-4:0)) #把时间尺度轴的标签显示为绝对值abs的形式,即把[-4,0]显示成[4,0]
2.2.2.6. 系统发育网(phylogenetic reticulum) —— geom_taxalink
系统发育网一般是在系统发育树绘制的基础上添加物种间的杂交和基因流关系。
1 | ggtree(tree) + geom_tiplab() |
2.2.2.7. 多棵图共同展示
- multiPhylo图
多棵树并列展示。
1
2
3
4trees <- read.newick("samples.trees") #读取newick格式的多棵树文件,一行一棵树
class(trees) <- "multiPhylo"
ggtree(trees) + facet_wrap(~.id, scale="free") + geom_tiplab() #分面展示,树的行列数量随机。
ggtree(trees) + facet_wrap(~.id, ncol=10) #分面展示,每行10棵。修改每棵树的标签
1
2lbs<-c("Tree #1"="atp1","Tree #2"="atp4","Tree #3"="atp6","Tree #4"="atp8","Tree #5"="atp9","Tree #6"="ccmB","Tree #7"="ccmC","Tree #8"="ccmFC","Tree #9"="ccmFN","Tree #10"="cob","Tree #11"="cox1","Tree #12"="cox2","Tree #13"="cox3","Tree #14"="matR","Tree #15"="mttB","Tree #16"="nad1","Tree #17"="nad2","Tree #18"="nad3","Tree #19"="nad4","Tree #20"="nad4L","Tree #21"="nad5","Tree #22"="nad6","Tree #23"="nad7","Tree #24"="nad9","Tree #25"="rpl10","Tree #26"="rpl16","Tree #27"="rpl2","Tree #28"="rpl5","Tree #29"="rps1","Tree #30"="rps10","Tree #31"="rps12","Tree #32"="rps19","Tree #33"="rps3","Tree #34"="rps4","Tree #35"="sdh3","Tree #36"="sdh4") # 线粒体基因组的36个基因名为标签
ggtree(trees) + geom_tiplab() + geom_treescale()+geom_nodelab() + facet_wrap(~.id,ncol=4,scale="free",labeller=as_labeller(lbs))
- DensityTree图
多棵树重叠展示。1
2trees <- read.tree(system.file("extdata/RAxML", "RAxML_bootstrap.H3", package="treeio")) #读取系统自带树文件RAxML_bootstrap.H3,包含100棵树。
ggdensitree(trees, alpha=.3, colour='steelblue') + geom_tiplab(size=3) + xlim(0, 45) #重叠多棵树。bootstrap值高的线条一致性,低的线条较乱。
2.2.2.8. 一个生成树的例子
1 | library(treeio) |
3. reference
- ggtree paper:https://besjournals.onlinelibrary.wiley.com/doi/full/10.1111/2041-210X.12628
- book_visualization of phylogenetic trees](https://yulab-smu.top/treedata-book/index.html
- ggtree tutorial:https://guangchuangyu.github.io/ggtree-book/chapter-ggtree.html
- treeio tutorial:https://guangchuangyu.github.io/ggtree-book/chapter-treeio.html
- facet_plot:https://guangchuangyu.github.io/2016/10/facet_plot-a-general-solution-to-associate-data-with-phylogenetic-tree/
- 不同树的绘制:https://www.codenong.com/js9fda2b82cedb/
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。


